
生物信息学 普通高等教育 “十二五”规划教材 生物信息学 Bioinformatics e00 第三章:序列比对原理
第三章:序列比对原理 普通高等教育 “十二五”规划教材 生物信息学 Bioinformatics

第一节序列比对相关概念 一、序列比对目的及定义 (一)序列比对目的 通过比较两条或多条序列之间是否具有足够的相似 性,从而判定它们之间是否具有同源性。 (二)序列比对定义 序列比对(sequence alignment).是运用某种特定的 数学模型或算法,找出两个或多个序列之间的最大匹 配碱基或残基数,比对的结果反映了算法在多大程度 上提供序列之间的相似性关系及它们的生物学特征
第一节 序列比对相关概念 一、序列比对目的及定义 (一)序列比对目的 通过比较两条或多条序列之间是否具有足够的相似 性,从而判定它们之间是否具有同源性。 (二)序列比对定义 序列比对(sequence alignment)是运用某种特定的 数学模型或算法,找出两个或多个序列之间的最大匹 配碱基或残基数,比对的结果反映了算法在多大程度 上提供序列之间的相似性关系及它们的生物学特征

二、序列比对类型 global alignment (一)序列比对分类 local alignment 双序列比对 多序列比对
二、序列比对类型 (一)序列比对分类 双序列比对 多序列比对 global alignment local alignment

(二)编辑距离 AGCACAC-A LUIII A-CACACTA Match(a,a)一一字符匹配。 Delete(a,)一从第一条序列删除一个字符,或者在第二条序列相应的位置插入空位。 Replace(a,b)一以第二条序列中的字符b替换第一条序列中的字符a,a≠b。 Insert((-,b)一一在第一条序列插入空位字符,或删除第二条序列中对应字符b。 通过编辑操作计算的两条序列的距离称为编辑距离
(二)编辑距离 通过编辑操作计算的两条序列的距离称为编辑距离
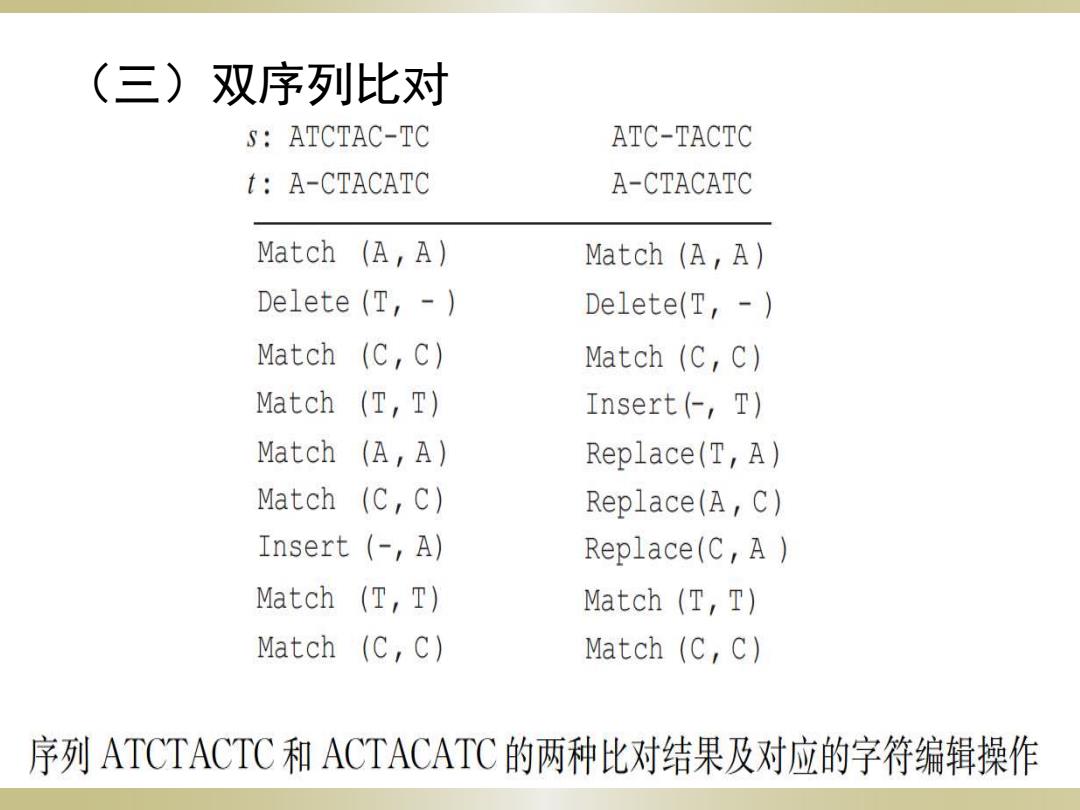
(三)双序列比对 s:ATCTAC-TC ATC-TACTC A-CTACATC A-CTACATC Match (A,A) Match (A,A) Delete (T,- Delete(T,- Match (C,C) Match (C,C) Match (T,T) Insert(←,T) Match (A,A) Replace(T,A) Match (C,C) Replace(A,C) Insert (-A) Replace(C,A Match (T,T) Match (T,T) Match (C,C) Match(C,c)】 序列ATCTACTC和ACTACATC的两种比对结果及对应的字符编辑操作
(三)双序列比对