
第一节:分子进化与系统发育 0八二、分子系统发育分析的基本概念 (三)物种树和基因/蛋白树 基于单个同源基因差异构建的系统发生树 真核生物域 细随域 古生菌域 动物 真菌 植物 其他细菌 蓝细菌 嗜泉古细菌 朊细菌 广域古细菌 类 生叶绿体的细菌 纤毛虫 其他单细胞 线粒体的细 真核生物 初生古生菊 超嗜热细菌 共同祖先 图9-2生命系统树
二、分子系统发育分析的基本概念 第一节:分子进化与系统发育 7 (三)物种树和基因/蛋白树 基于单个同源基因差异构建的系统发生树

第二节:分子系统发育树的构建方法 0似分子系统发育树的构建方法 利用生物大分子数据重建系统进化树,目前最常用的有4种方法 距离法、最大简约法、最大似然法和贝叶斯法。 最大简约法主要适用于序列相似性很高的情况; 距离法在序列具有比较高的相似性时适用; 最大似然法和贝叶斯法可用于任何相关的数据序列集合。 从计算速度来看,距离法的计算速度最快,其次是最大简约法和贝 叶斯方法,然后是最大似然法。 8
分子系统发育树的构建方法 第二节:分子系统发育树的构建方法 8 利用生物大分子数据重建系统进化树,目前最常用的有4种方法 距离法、最大简约法、最大似然法和贝叶斯法。 最大简约法主要适用于序列相似性很高的情况; 距离法在序列具有比较高的相似性时适用; 最大似然法和贝叶斯法可用于任何相关的数据序列集合。 从计算速度来看,距离法的计算速度最快,其次是最大简约法和贝 叶斯方法,然后是最大似然法
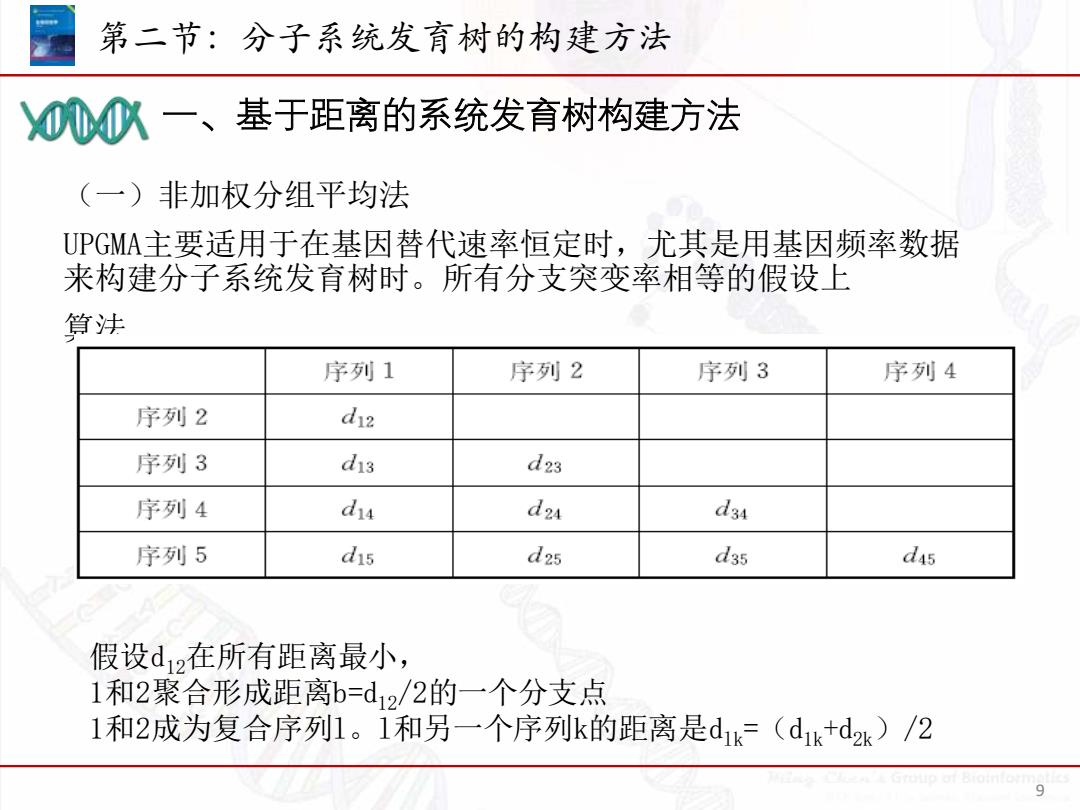
第二节:分子系统发育树的构建方法 例心一、基于距离的系统发育树构建方法 (一)非加权分组平均法 UPGMA主要适用于在基因替代速率恒定时,尤其是用基因频率数据 来构建分子系统发育树时。所有分支突变率相等的假设上 算注 序列1 序列2 序列3 序列4 序列2 di2 序列3 d13 d23 序列4 d14 d24 d34 序列5 d15 d2s d35 d45 假设d12在所有距离最小, 1和2聚合形成距离b=d2/2的一个分支点 1和2成为复合序列1.1和另一个序列k的距离是d1k=(d1k+d2w)/2 9
一、基于距离的系统发育树构建方法 第二节:分子系统发育树的构建方法 9 (一)非加权分组平均法 UPGMA主要适用于在基因替代速率恒定时,尤其是用基因频率数据 来构建分子系统发育树时。所有分支突变率相等的假设上 算法 假设d12在所有距离最小, 1和2聚合形成距离b=d12/2的一个分支点 1和2成为复合序列l。l和另一个序列k的距离是dlk=(d1k+d2k)/2
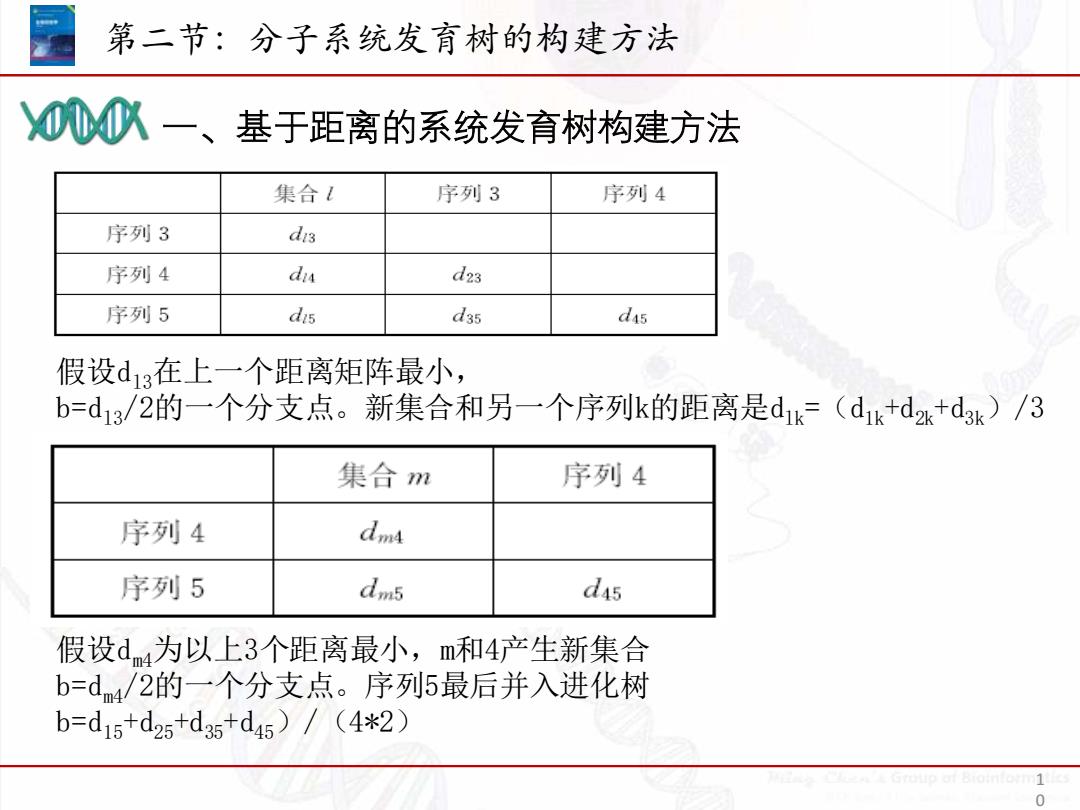
第二节:分子系统发育树的构建方法 例队一、基于距离的系统发育树构建方法 集合1 序列3 序列4 序列3 dis 序列4 di d23 序列5 dis d35 d45 假设d3在上一个距离矩阵最小, b=d13/2的一个分支点。新集合和另一个序列k的距离是d1k(d1k+d2+dk)/3 集合m 序列4 序列4 dmA 序列5 dm5 d45 假设d4为以上3个距离最小,m和4产生新集合 b=d4/2的一个分支点。序列5最后并入进化树 b=d15+d25+d35+d45)/(4*2) 1 0
一、基于距离的系统发育树构建方法 第二节:分子系统发育树的构建方法 1 0 假设dl3在上一个距离矩阵最小, b=dl3/2的一个分支点。新集合和另一个序列k的距离是dlk=(d1k+d2k+d3k)/3 假设dm4为以上3个距离最小,m和4产生新集合 b=dm4/2的一个分支点。序列5最后并入进化树 b=d15+d25+d35+d45)/(4*2)

第二节:分子系统发育树的构建方法 0心一、基于距离的系统发育树构建方法 集合L 序列3 序列3 di 集合m dim d3m d13不是最小,d4s才是,4和5成为新集合,则b4s/2,新的矩阵如图,dm (d35+d4s)/2 d1m(d14+d15+d24+d25)/4 1 1
一、基于距离的系统发育树构建方法 第二节:分子系统发育树的构建方法 1 1 dl3不是最小,d45才是,4和5成为新集合,则b45/2,新的矩阵如图,d3m = (d35+d45)/2 dlm=(d14+d15+d24+d25)/4