
3. 限制酶的命名☆ 寄主菌属名的第一个字母和种名的头两个字母组成3个斜体字母的 略语,表示酶来源的菌种名称,如大肠杆菌Escherichia coli表示为 Eco,流感嗜血菌Haemophilus influenzae表示为Hin; 用一个正体字母表示菌株的类型,比如EcoR; 用罗马数字表示从特殊菌株中分离的第几个限制性酶,比如EC0RI、 HindⅢ
3. 限制酶的命名 寄主菌属名的第一个字母和种名的头两个字母组成3个斜体字母的 略语,表示酶来源的菌种名称,如大肠杆菌Escherichia coli 表示为 Eco , 流感嗜血菌Haemophilus influenzae 表示为Hin; 用一个正体字母表示菌株的类型,比如Eco R; 用罗马数字表示从特殊菌株中分离的第几个限制性酶,比如Eco R I、 Hind III

属名 种名 株名 Haemophilus influenzae d嗜血流感杆菌d株 Hind III Hind III 同一菌株中所含的多个不同的限制性核酸内切酶

4.限制性酶的种类 亚单位组成, 识别序列的种类,是否需要辅助因子 主要特性 1型 川型 川型 限制修饰 多功能 单功能 双功能 蛋白结构 异源三聚体 同源二聚体 异源二聚体 辅助因子 ATP Mg2+SAM Mg2+ ATP Mg2+SAM 识别序列 TGAN&TGCT 旋转对称序列 GAGCC AACNGTGC CAGCAG 切割位点 距识别序列1kb处 识别序列内或附近 距识别序列下游 随机性切割 特异性切割 24-26bp处
亚单位组成, 识别序列的种类, 是否需要辅助因子 4. 限制性酶的种类

type lI 93% 1、识别位点的特异性:每种酶都有其特定的DNA识别位点, 通常是由4、5、6或7个核苷酸组成的特定序列(靶序列)。 2、识别序列的对称性:靶序列通常具有双重旋转对称的结构, 即双链的核苷酸顺序呈回文结构。 3、切割位点的规范性:双链DNA被酶切后,分布在两条链上 的切割位点旋转对称(可形成粘性末端或平末端的DNA分子)
type II 93% 1、识别位点的特异性:每种酶都有其特定的DNA识别位点, 通常是由4、5、6或7个核苷酸组成的特定序列(靶序列)。 2、识别序列的对称性:靶序列通常具有双重旋转对称的结构, 即双链的核苷酸顺序呈回文结构。 3、切割位点的规范性:双链DNA被酶切后,分布在两条链上 的切割位点旋转对称(可形成粘性末端或平末端的DNA分子)
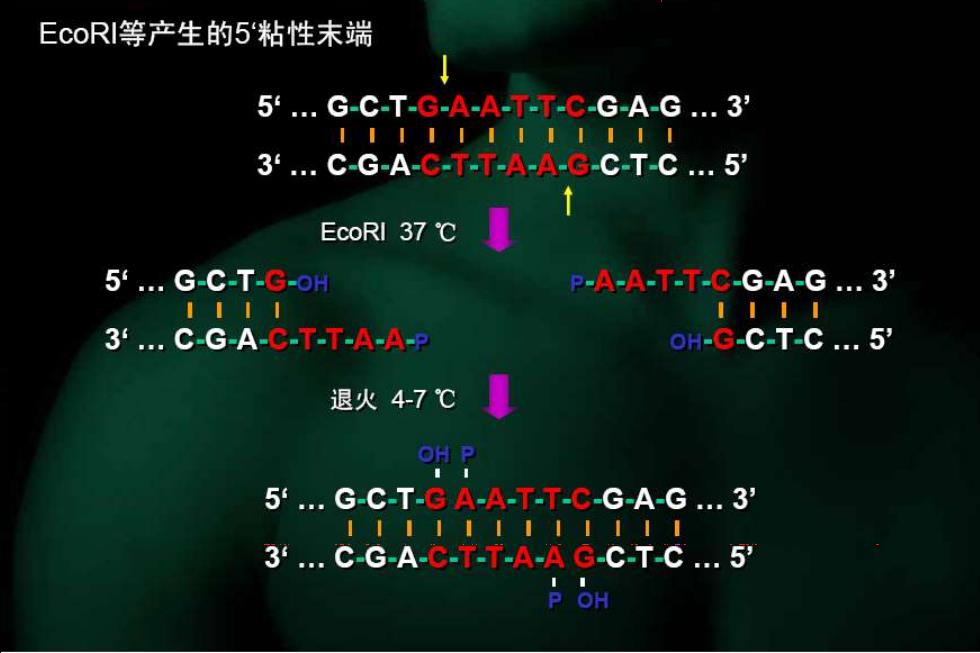
EcoRI等产生的5'粘性未端 5.G-C-T-G-A-A-T-T-C-G-A-G.3' I IIIIIIIIIII 3.C-G-A-C-T-T-A-A-G-C-T-C.5' EcoRI37℃ 5.G-C-T-G-OH P-A-A-T-T-C-G-A-G.3' 1111 1111 3.C-G-A-C-T-T-A-A-9 ©H-G-C-T-C.5 退火4-7℃ OH P 5.G-C-T-G A-A-T-T-C-G-A-G.3' 111111111111 3.C-G-A-C-T-T-A-A G-C-T-C.5 P OH