
第二节:试验设计和测序流程 00 、测序流程 目标样本 RNA分离纯化 打断,构建cDNA文库, MMMN 长度筛选,添加接头 MANononmnNo 肿瘤组织 正常组织 MMMMMMM mRNA:Poly A富集 Poy(A)尾 ncRNA:rRNA移除 上机测序 比对到参考基因组或转录组 内含子 RNA前体 外显子 未测序RNA RNA读段 转录本 短读段 可变剪切区域 短插入片段 Griffith,M.(2015)PLoS computational biology
第二节:试验设计和测序流程 7 Griffith, M. (2015) PLoS computational biology mRNA:Poly A富集 ncRNA:rRNA移除 测序流程
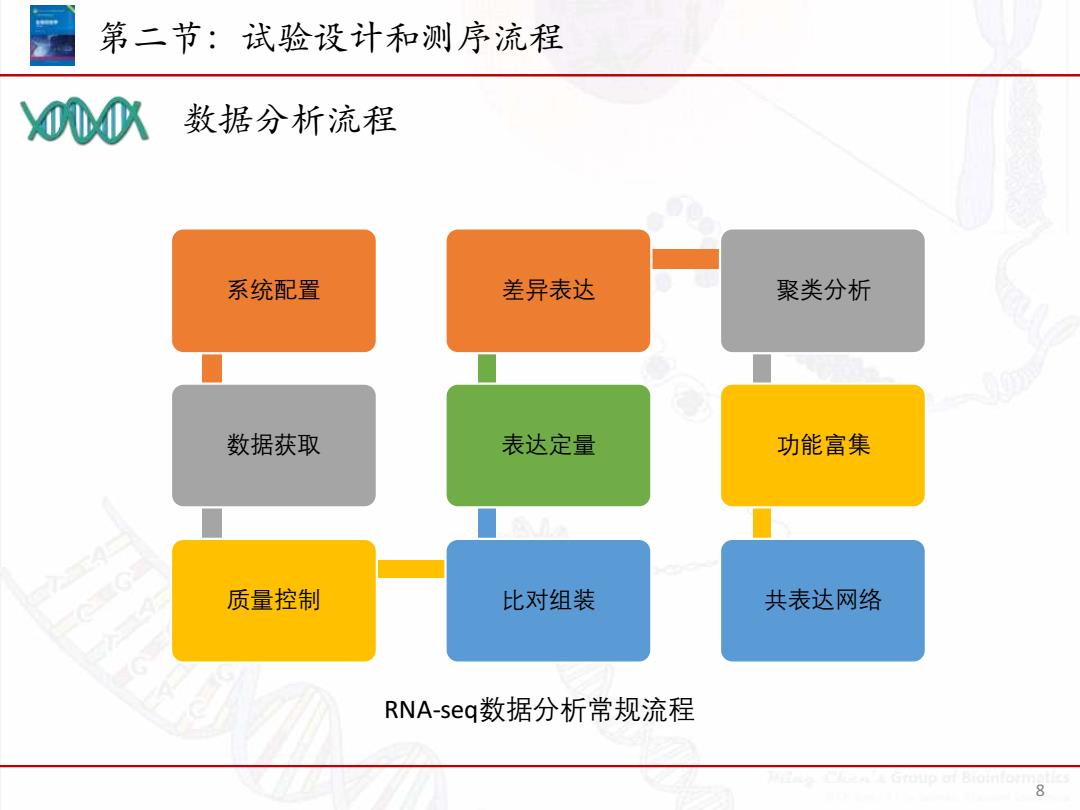
第二节:试验设计和测序流程 000八 数据分析流程 系统配置 差异表达 聚类分析 数据获取 表达定量 功能富集 质量控制 比对组装 共表达网络 RNA-seq数据分析常规流程 8
第二节:试验设计和测序流程 8 系统配置 数据获取 质量控制 比对组装 表达定量 差异表达 聚类分析 功能富集 共表达网络 RNA-seq数据分析常规流程 数据分析流程

第二节:试验设计和测序流程 00M八 系统配置 8L△nux Microsoft Java Sun Microsystems R 语言基础 080+ Public Galaxy Servers and stiff counting 9
第二节:试验设计和测序流程 9 系统配置

第三节:转录组数据核心分析 00 、数据获取 GSA THE CANCER GENOME ATLAS NIH National Cancer Institute National Human Genome Research Institute Genome Sequence Archive NCBI SRA TCGA/GDC(cancer) ArrayExpress NIH NATIONAL CANCER INSTITUTE Genomic Data Commons fastq-dump EBI ArrayExpress (SRAToolkit) 公共数据库 测序公司 Fastq文件格式: esRR3418005,1HAL:1262:D2 EWTACXX:8:1101:1602:21361 ength=100 GGCAAGATCTGATCTCTCAGCAACTCAATTACAACCATAACCGCGTGTGACTTCTAAGCC +SRR3418005,1HAL:1282:D2 EWTACXX:8:1101:1602:21361 ength-100 ::1DDAD?DDFHFGHII EIIIGHHHDHGIGDFGHGGEGIIHGBEDHEEGGFIDEEAAEH Q8RR3418005.2HAL:1282:D2 EWTACXX:8:1101:1550:21931 ength=100 ATCTGATTCAATCATAAATTTTACACAATCAATTTGTCGGTACTCTCCTTTTGGTCATAT +sRR3418005,2HAL:12B2:D2 EWTACXX:8:1101:1550:21931 ength=100 <?0BDD:DCDDHHGIBGA<FFF<:FDGGCE9CADHIEG:?DF):BBB9??BGG60?B<B- @sRR3418005,3HAL:1282:D2 EWTACXX:8:1101:1632:22051 ength-100 AGACGCTCGTACCAAATCCGTTACCGTCTCCGTCGTTACCTCCTCCTTCGCGACGGGAAC +sRR3418005.3HAL:12B2:D2 EWTACXX:8:1101:1632:22051 ength=100 --+:A@DDE@F8C<A+CBEFIIFIF@DFFFFFDF(@F--.-70FEF).-@A## esRR3418005.4HAL:1282:D2 EWTACXX:8:1101:1588:22271 ength=100 CTCATTTTTATTACCGCATATATGACATATGATCAATTACATAAAGAAGCAAATCTTAGO +SRR3418005,4HAL:12B2:D2 EWTACXX:8:1101:158B:22271 ength-100 30-DDDAFHFAHEH?FF<EBF9EHDHHGOACCGICHHCHGIEHG<DFB9DDFHEGFHDG 0sRR3418005.5HAL:1282:D2 EWTACXX:8:1101:1991:21131 ength=100 CGGAAGCAGCTGAGAAGCCTCATGGTTACCAACAAGAGCATCCTCATCAGTTNCACCATA +9RR3418005.5HAL:1282:D2 EWTACXX:8:1101:1991:21131 ength=100 GCOFFFDFHHHFGIIGIGIJJJJIJIIIIIIJJJJCEIGIIJHIJEIIJJHI#-<FFHIJ 10
第三节:转录组数据核心分析 10 Fastq文件格式: NCBI SRA EBI ArrayExpress TCGA/GDC(cancer) fastq-dump (SRAToolkit) 公共数据库 测序公司 数据获取

第三节:转录组数据核心分析 00M队 质量控制 去接头;过滤 低质量reads FastQC-测序质量评估 FASTX-Toolkit,Trimmomatic--一质量控制 eFastQC Report Summary Per base sequence quality Basic Statistics Quality scores across all bases (Sanger /lllumina 1.9 encoding) 40 Per base sequence quality 3 Per tile sequence quality Per sequence quality scores 34 Per base sequence content Per sequence GC content ⑦Per base N content Sequence Length Distribution 66420 Sequence Duplication Levels Overrepresented sequences ①Adapter Content ⑧Kmer Content 64120 8 6 4 2 0 12345678912-1318-1924-2530-3136-3742-4348-4954-55606166-6772-7378-79B4-85909196-97 Position in read(bp》 11
第三节:转录组数据核心分析 11 FastQC—测序质量评估 FASTX-Toolkit,Trimmomatic—质量控制 质量控制 去接头;过滤 低质量reads