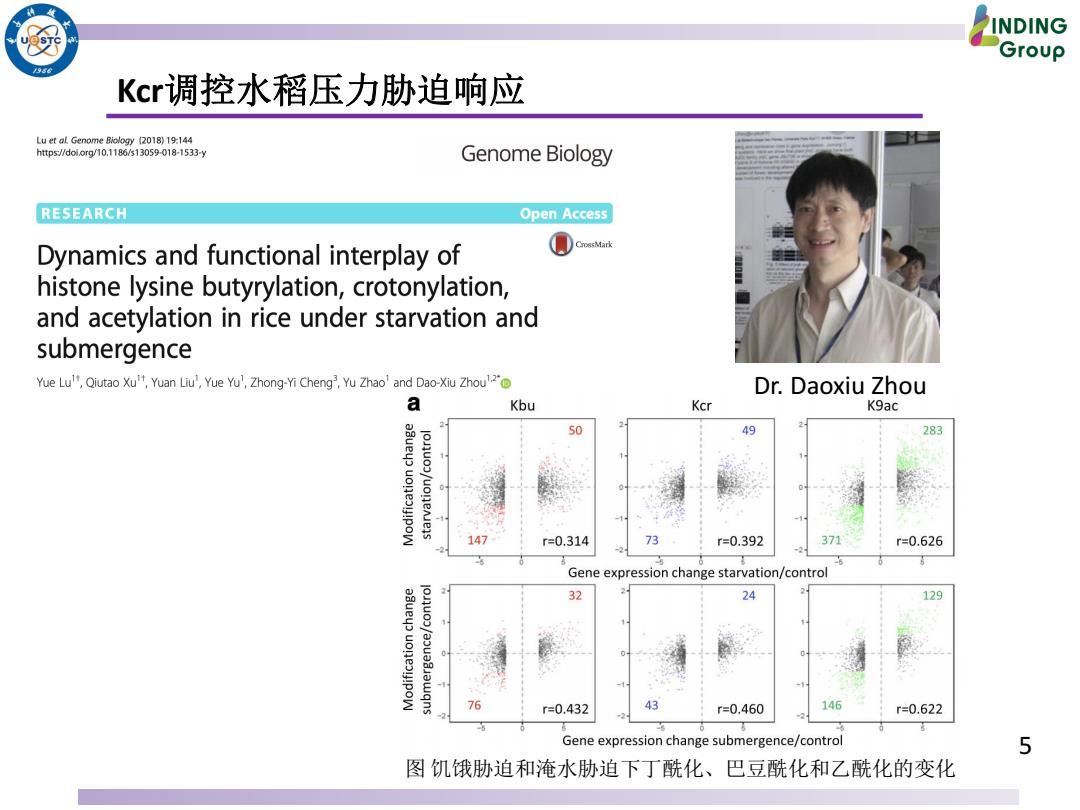
INDING Group Kcr调控水稻压力胁迫响应 Lu et aL Genome Biology (2018)19:144 http5/doi.org/10,1186/513059-018-1533-y Genome Biology RESEARCH Open Access Dynamics and functional interplay of histone lysine butyrylation,crotonylation, and acetylation in rice under starvation and submergence Yue Lut,Qiutao Xu't,Yuan Liu',Yue Yu',Zhong-Yi Cheng3,Yu Zhao'and Dao-Xiu Zhou' Dr.Daoxiu Zhou a Kbu Kcr K9ac 50 49 283 147 r=0.314 73 r=0.392 371 r=0.626 Gene expression change starvation/control 32 24 129 76 r=0.432 43 r=0.460 146 r=0.622 Gene expression change submergence/control 5 图饥饿胁迫和淹水胁迫下丁酰化、巴豆酰化和乙酰化的变化
5 Kcr调控水稻压力胁迫响应 图 饥饿胁迫和淹水胁迫下丁酰化、巴豆酰化和乙酰化的变化 Dr. Daoxiu Zhou
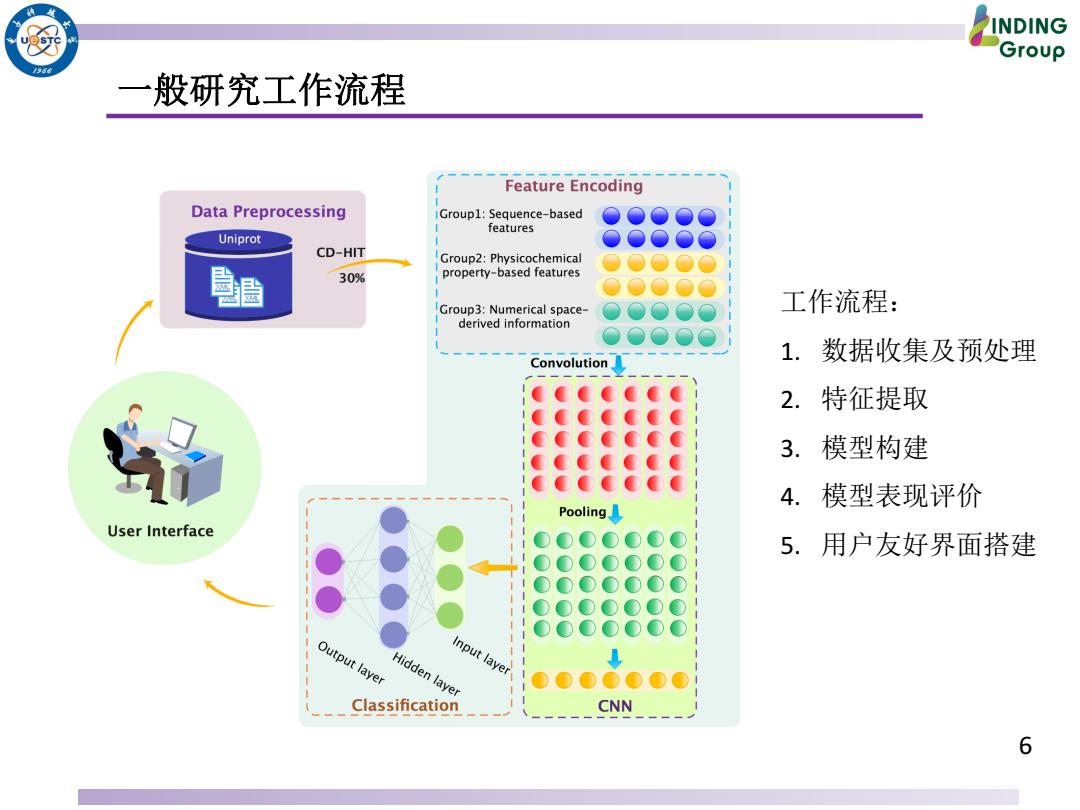
INDING Group 一般研究工作流程 Feature Encoding Data Preprocessing Groupl:Sequence-based features Uniprot CD-HIT Group2:Physicochemical 30% property-based features Group3:Numerical space- 工作流程: derived information 1.数据收集及预处理 Convolution 2.特征提取 3.模型构建 4.模型表现评价 Pooling User Interface 5.用户友好界面搭建 Output layer Hidden layer Input layer Classification CNN 6
6 一般研究工作流程 工作流程: 1. 数据收集及预处理 2. 特征提取 3. 模型构建 4. 模型表现评价 5. 用户友好界面搭建
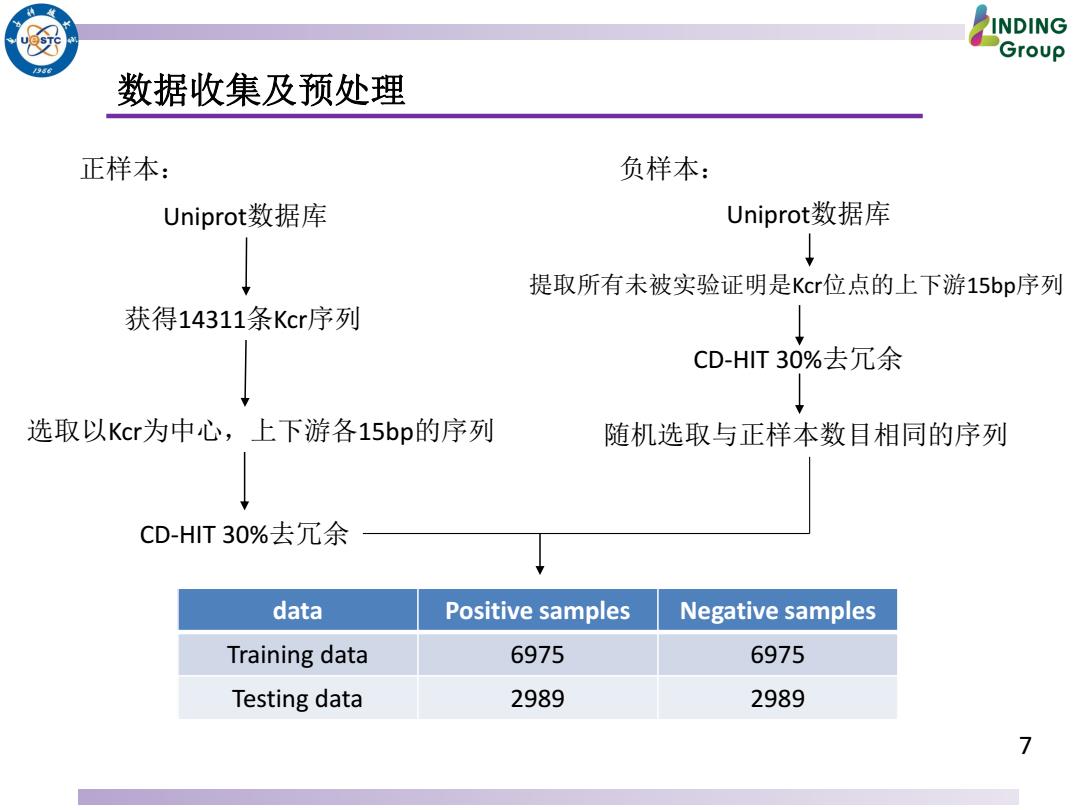
INDING Group 数据收集及预处理 正样本: 负样本: Uniprot数据库 Uniprot数据库 提取所有未被实验证明是Kcr位点的上下游15bp序列 获得14311条Kcr序列 CD-HIT30%去冗余 选取以Kcr为中心,上下游各15bp的序列 随机选取与正样本数目相同的序列 CD-HIT30%去冗余 data Positive samples Negative samples Training data 6975 6975 Testing data 2989 2989 7
7 数据收集及预处理 Uniprot数据库 获得14311条Kcr序列 选取以Kcr为中心,上下游各15bp的序列 CD-HIT 30%去冗余 data Positive samples Negative samples Training data 6975 6975 Testing data 2989 2989 正样本: 负样本: Uniprot数据库 提取所有未被实验证明是Kcr位点的上下游15bp序列 CD-HIT 30%去冗余 随机选取与正样本数目相同的序列

ov 96 特征提取 》基于序列信息的特征 》基于物化性质的特征 》基于数值空间信息的特征 8
8 特征提取 基于序列信息的特征 基于物化性质的特征 基于数值空间信息的特征

INDING Group 基于序列信息的特征 1.K-space氨基酸对组分(CKSAAP) CKSAAP反映了氨基酸对之间的短程相互作用信息。假设K=0,那么有400(20*20) 个间隔为0的残基对(即AA,AC,AD,,YY).可以用下式计算特征向量: NAA NAC NAP NYY NTotal'NTotal'NTotal 'NTotal/400 其中,NTotau是总的残基组分的长度(例如,如果长度为L的蛋白质片段为31且 k=0、1、2、3、4和5,则NTotal=L-k-1,分别对应为为30、29、28、27、 26和25)。NAA、W4c、NAD、Nyy分别代表氨基酸对的频率。在本研究中设k= 0、1、2、3、4和5,因此基于CKSAAP的特征向量的总维数为400×6=2400。 9
9 基于序列信息的特征 1. K-space氨基酸对组分 (CKSAAP) CKSAAP反映了氨基酸对之间的短程相互作用信息。假设K = 0, 那么有400 (20*20) 个间隔为0的残基对 (即AA, AC, AD, …, YY). 可以用下式计算特征向量: 𝑁𝐴𝐴 𝑁𝑇𝑜𝑡𝑎𝑙 , 𝑁𝐴𝐶 𝑁𝑇𝑜𝑡𝑎𝑙 , 𝑁𝐴𝐷 𝑁𝑇𝑜𝑡𝑎𝑙 , ⋯ , 𝑁𝑌𝑌 𝑁𝑇𝑜𝑡𝑎𝑙 400 其中, 𝑁𝑇𝑜𝑡𝑎𝑙是总的残基组分的长度(例如,如果长度为 𝐿 的蛋白质片段为31且 k = 0、1、2、3、4和5,则𝑁𝑇𝑜𝑡𝑎𝑙 = 𝐿 − 𝑘 − 1,分别对应为为30、29、28、27、 26和25)。 𝑁𝐴𝐴 、𝑁𝐴𝐶 、𝑁𝐴𝐷 、𝑁𝑌𝑌分别代表氨基酸对的频率。 在本研究中设𝑘 = 0、1、2、3、4和5,因此基于CKSAAP的特征向量的总维数为400×6 = 2400