
南京农业大学学报2018,41(2):240-247 http://nauxb.njau.edu.cn Journal of Nanjing Agricultural University D0I:10.7685/jnau.201705022 可 张飞雪,虞章红,刘坤宇,等.不结球白菜醛酮还原酶Bc4KR4C9基因的克隆及表达分析[J].南京农业大学学报,2018,41(2):240-247 不结球白菜醛酮还原酶BCAKR4C9基因的 克隆及表达分析 张飞雪,虞章红,刘坤宇,文锴,王建军,侯喜林,李英* (南京农业大学园艺学院/作物遗传与种质创新国家重点实验室/农业部华东地区园艺作物生物学 与种质创新重点实验室,江苏南京210095) 摘要:[目的]本文旨在探究醛酮还原酶AKR4C亚家族BAKR4C9基因在不同激素处理及逆境胁迫中的表达模式,从而分 析BAKR4C9基因在不结球白菜作物中的生长代谢和逆境胁迫中可能发挥的作用。[方法]以不结球白菜·苏州青'为材 料,克隆了BeAKR4C9基因的全长,应用生物信息学分析了其氨基酸序列,并通过实时荧光定量PCR技术分析BAKR4C9基 因在不同组织,高温、低温、NC胁迫、伤害胁迫,以及水杨酸、茉莉酸甲酯和脱落酸等处理下的基因表达变化。[结果]序 列分析表明,BcAKR-4C9基因包含1个长度为948bp的开放式阅读框(ORF),编码315个氨基酸。氨基酸序列多重比对和 进化树分析表明,BcAKR4C9与其他植物AKR4C9基因同源性较高,具有高度保守性。荧光定量PCR分析表明,BeAKR4C9 在叶片中的表达量高于根中,高温和低温处理下表达上调,且具有相似的表达模式。在0-20 g-L-NaCl质量浓度范围内基 因相对表达量随NC1质量浓度的升高而升高,伤害处理后能够迅速产生应激响应,并能够被水杨酸、茉莉酸甲酯和脱落酸 诱导表达。[结论]不结球白菜BAKR4C9基因能够被不同激素诱导表达,并且参与到多种逆境胁迫响应。 关键词:不结球白菜:醛酮还原酶AKR4C亚家族:序列分析:非生物胁迫 中图分类号:S634.3 文献标志码:A 文章编号:1000-2030(2018)02-0240-08 Cloning and expression analysis of aldo-keto reductase gene BcAKR4C9 from non-heading Chinese cabbage ZHANG Feixue,YU Zhanghong,LIU Kunyu,WEN Kai,WANG Jianjun,HOU Xilin,LI Ying' College of Horticulture/State Key Laboratory of Crop Genetics and Germplasm Enhancement/ Key Laboratory of Biology and Germplasm Enhancement of Horticultural Crops in East China, Ministry of Agriculture,Nanjing Agricultural University,Nanjing 210095,China) Abstract:[Objectives]The purpose of this study was to explore the expression pattern of BeAKR4C9 gene of AKR4C subfamily under different hormone treatments and stress conditions,so as to analyze the possible role of BcAKR4C9 gene in growth development and stress in non-heading Chinese cabbage.Methods]The whole cDNA sequence BcAKR4C9 gene was cloned from non-heading Chinese cabbage'Suzhouqing',and its amino acid sequence was analyzed by bioinformatics,and real-time fluorescence quantitative PCR technology was used to explore its expression pattern under different tissues,different stress conditions(high temperature,low temperature,NaCl stress,wound stress)and different hormone treatments (salicylic acid,methyl jasmonate and abscisic acid). [Results]The sequence analysis showed that the BeAKR4C9 gene contained an open reading frame(ORF)with a length of 948 bp, encoding 315 amino acids.The amino acid sequence multiple alignment and mapping phylogenetic tree showed that BcAKR4C9 gene was highly homologous to the AKR4C9 genes from other plants and highly conserved.Real-time fluorescence quantitative PCR analy- sis showed that the expression of BcAKR4C9 gene in the leaves was higher than that in the roots,and it was up-regulated at high temperature and low temperature and had similar expression patterns.Its expression increased with the increase of salt concentration from 0 to 20 gL.The expression of BeAKR4C9 also could be induced by salicylic acid,methyl jasmonate and abscisic acid. Conclusions]The non-heading Chinese cabbage BcAKR4C9 gene can be induced by different hormones and is involved in a variety of stress responses. Keywords:non-heading Chinese cabbage;aldo-keto reductase AKR4C subfamily;sequence analysis;abiotic stress 收稿日期:2017-05-15 基金项目:国家自然科学基金项目(31471886):国家973计划子课题项目(2009CB119001-04):江苏省农业科技支撑项目(BE2012325) 作者简介:张飞雪,硕士研究生。通信作者:李英,教授,研究方向为蔬菜分子生物学与遗传育种,E-mail:yingli(@jau.d血.cm
南京农业大学学报 2018ꎬ41(2):240-247 http: / / nauxb.njau.edu.cn Journal of Nanjing Agricultural University DOI:10.7685 / jnau.201705022 收稿日期:2017-05-15 基金项目:国家自然科学基金项目(31471886)ꎻ国家 973 计划子课题项目(2009CB119001-04)ꎻ江苏省农业科技支撑项目(BE2012325) 作者简介:张飞雪ꎬ硕士研究生ꎮ ∗ 通信作者:李英ꎬ教授ꎬ研究方向为蔬菜分子生物学与遗传育种ꎬE ̄mail:yingli@ njau.edu.cnꎮ 张飞雪ꎬ虞章红ꎬ刘坤宇ꎬ等. 不结球白菜醛酮还原酶 BcAKR4C9 基因的克隆及表达分析[J]. 南京农业大学学报ꎬ2018ꎬ41(2):240-247. 不结球白菜醛酮还原酶 BcAKR4C9 基因的 克隆及表达分析 张飞雪ꎬ虞章红ꎬ刘坤宇ꎬ文锴ꎬ王建军ꎬ侯喜林ꎬ李英∗ (南京农业大学园艺学院/ 作物遗传与种质创新国家重点实验室/ 农业部华东地区园艺作物生物学 与种质创新重点实验室ꎬ江苏 南京 210095) 摘要:[目的]本文旨在探究醛酮还原酶 AKR4C 亚家族 BcAKR4C9 基因在不同激素处理及逆境胁迫中的表达模式ꎬ从而分 析 BcAKR4C9 基因在不结球白菜作物中的生长代谢和逆境胁迫中可能发挥的作用ꎮ [方法]以不结球白菜‘苏州青’为材 料ꎬ克隆了 BcAKR4C9 基因的全长ꎬ应用生物信息学分析了其氨基酸序列ꎬ并通过实时荧光定量 PCR 技术分析 BcAKR4C9 基 因在不同组织ꎬ高温、低温、NaCl 胁迫、伤害胁迫ꎬ以及水杨酸、茉莉酸甲酯和脱落酸等处理下的基因表达变化ꎮ [结果]序 列分析表明ꎬBcAKR4C9 基因包含 1 个长度为 948 bp 的开放式阅读框(ORF)ꎬ编码 315 个氨基酸ꎮ 氨基酸序列多重比对和 进化树分析表明ꎬBcAKR4C9 与其他植物 AKR4C9 基因同源性较高ꎬ具有高度保守性ꎮ 荧光定量 PCR 分析表明ꎬBcAKR4C9 在叶片中的表达量高于根中ꎬ高温和低温处理下表达上调ꎬ且具有相似的表达模式ꎮ 在 0~ 20 gL -1NaCl 质量浓度范围内基 因相对表达量随 NaCl 质量浓度的升高而升高ꎬ伤害处理后能够迅速产生应激响应ꎬ并能够被水杨酸、茉莉酸甲酯和脱落酸 诱导表达ꎮ [结论]不结球白菜 BcAKR4C9 基因能够被不同激素诱导表达ꎬ并且参与到多种逆境胁迫响应ꎮ 关键词:不结球白菜ꎻ醛酮还原酶 AKR4C 亚家族ꎻ序列分析ꎻ非生物胁迫 中图分类号:S634.3 文献标志码:A 文章编号:1000-2030(2018)02-0240-08 Cloning and expression analysis of aldo ̄keto reductase gene BcAKR4C9 from non ̄heading Chinese cabbage ZHANG FeixueꎬYU ZhanghongꎬLIU KunyuꎬWEN KaiꎬWANG JianjunꎬHOU XilinꎬLI Ying ∗ (College of Horticulture / State Key Laboratory of Crop Genetics and Germplasm Enhancement / Key Laboratory of Biology and Germplasm Enhancement of Horticultural Crops in East Chinaꎬ Ministry of AgricultureꎬNanjing Agricultural UniversityꎬNanjing 210095ꎬChina) Abstract:[Objectives] The purpose of this study was to explore the expression pattern of BcAKR4C9 gene of AKR4C subfamily under different hormone treatments and stress conditionsꎬso as to analyze the possible role of BcAKR4C9 gene in growth development and stress in non ̄heading Chinese cabbage. [Methods] The whole cDNA sequence BcAKR4C9 gene was cloned from non ̄heading Chinese cabbage‘Suzhouqing’ꎬand its amino acid sequence was analyzed by bioinformaticsꎬand real ̄time fluorescence quantitative PCR technology was used to explore its expression pattern under different tissuesꎬdifferent stress conditions( high temperatureꎬlow temperatureꎬNaCl stressꎬ wound stress) and different hormone treatments ( salicylic acidꎬ methyl jasmonate and abscisic acid). [Results]The sequence analysis showed that the BcAKR4C9 gene contained an open reading frame(ORF)with a length of 948 bpꎬ encoding 315 amino acids. The amino acid sequence multiple alignment and mapping phylogenetic tree showed that BcAKR4C9 gene was highly homologous to the AKR4C9 genes from other plants and highly conserved. Real ̄time fluorescence quantitative PCR analy ̄ sis showed that the expression of BcAKR4C9 gene in the leaves was higher than that in the rootsꎬand it was up ̄regulated at high temperature and low temperature and had similar expression patterns. Its expression increased with the increase of salt concentration from 0 to 20 gL -1 . The expression of BcAKR4C9 also could be induced by salicylic acidꎬmethyl jasmonate and abscisic acid. [Conclusions]The non ̄heading Chinese cabbage BcAKR4C9 gene can be induced by different hormones and is involved in a variety of stress responses. Keywords:non ̄heading Chinese cabbageꎻaldo ̄keto reductase AKR4C subfamilyꎻsequence analysisꎻabiotic stress

第2期 张飞雪,等:不结球白菜醛酮还原酶BcAKR4C9基因的克隆及表达分析 241 在植物生长发育的各个阶段,植物会不可避免地遇到一些生物和非生物胁迫山。研究表明:植物在 遭遇非生物胁迫时,活性氧与活性乙醛水平增加,导致植物的胁迫诱导损伤),为了清除植物体内产生的 活性氧、活性乙醛等有毒物质,植物会利用大量具有氧化还原活性的酶类使有毒物质分解为毒性较小的乙 醇和碳水化合物3),而醛酮还原酶(AKR)家族通常催化醛、酮、类固醇以及其他羰基化合物的还原)。 AKR家族在逆境防御中起重要作用,能够被多种逆境胁迫诱导4刊:在植物中它们起着反应性醛解毒、渗 透压生成和次级代谢物合成的关键作用8】。 AKR家族,是以NADP(H)为辅酶而催化一系列醛酮底物发生氧化还原反应,具有经典的(a/B)8桶 状折叠结构,在进化上高度保守。其中,(a/B)8桶状结构羧基端的3个环(LooP A、LooP B、LooP C)的不 同组成和大小决定了其底物的特异性)。AKR催化活性位点是由N末端(Asp50、Tyr55、Lys84)所构 成[),催化四联体(Asp50、Ty55、Lys84、His117)被认为是醛酮还原酶负责底物催化的关键氨基酸1o);另 外,发现C末端形成的高度保守的氨基酸残基(Ser271、Arg276)在辅因子结合中起重要作用。 AKR4C亚家族与植物的耐受性相关[)。AKR4C能够被高C0,浓度和高光照下诱导表达,使细胞内 光合速率增强[山。在单子叶植物中首先鉴定到的AKR4C1被认为是耐旱性相关的蛋白并能够被脱落酸 诱导表达12】。AKR4C9为AKR4C亚家族的成员,在逆境胁迫中能够起到醛酮解毒的作用1],被认为是保 护植物免受应激氧化胁迫的重要的候选基因)。在大麦中过表达拟南芥AKR4C9基因可以在氧化胁迫和 镉胁迫中增强其醛酮解毒反应,转基因植株呼吸速率降低,叶绿素含量增加并且光合速率提高):山梨醇 浓度相对于野生型提高2~4倍,其耐寒性和再生能力得到显著提高[4)。 本研究以不结球白菜品种·苏州青'为试验材料,克隆了AKR4C9同源基因,并且进行了相关的生物 信息学分析。利用实时荧光定量PCR技术分别测定了高温、低温、NaCl、伤害处理下的基因表达水平,以 及在脱落酸、茉莉酸甲酯和水杨酸激素处理下基因的表达变化,旨在进一步了解BAKR4C9基因在不结球 白菜中的功能。 1材料与方法 1.1材料及处理 供试材料为不结球白菜(Brassica campestris ssp.chinensis Makino)品种‘苏州青',由南京农业大学园艺学 院不结球白菜课题组提供。将‘苏州青'种植于南京农业大学人工气候室,在植株4叶1心期时,选取生长整 齐一致的幼苗作为试验材料。以42℃热激和4℃低温处理幼苗,处理设备为RXZ智能型人工气候培养箱, 光照强度为100molm2s,处理后0、4、6、8和24h取样,每处理10株,3次重复。在每片叶上打3个孔 (保留圆片不致脱离,孔径大小为1cm)作为伤害处理,未打孔的作为对照,分别在0、4、8、12和24h后取样, 每处理10株,3次重复。分别以5、10、15、20、25g·L的NaC溶液喷洒叶片背面,以清水为对照,然后用透光 塑料罩罩住,处理12h后取样,每处理10株,3次重复。选用2 mmol.L-水杨酸(SA)、50molL菜莉酸甲 酯(JA)和50molL脱落酸(ABA)溶液分别喷施4叶1心期的叶片,然后用透光塑料罩罩住,处理时间为 0、4、8、12和24h,每处理10株,3次重复。材料收集后液氮速冻,存放-80℃保存备用。 1.2RNA的提取与cDNA的合成 RNA提取试剂盒购自天根生化科技有限公司,具体方法参照RpPureNApre植物总RNA提取试剂盒说明 书。用‘苏州青'叶片的总RNA为模板,按照PrimeScript RT试剂盒(TaKaRa)说明书反转录合成cDNA。 1.3不结球白菜BcAKR-4C9基因的克隆 根据大白菜数据库(htp:/brassicadb.org/brad/searchGene.php)的基因序列编号Bra0000I8,利用 Primer Premier6.0软件设计1对I物,引物分别为AKR4C9-F:ATGGCTAACGCAATTAGAT,AKR4C9-R: TTATATCTCGCCATCCCATAAGT。以不结球白菜‘苏州青'叶片cDNA作为模板进行PCR反应,反应体系 按照TaKaRa公司的rTag酶说明书进行。反应程序为:95℃5min:95℃30s,58℃30s,72℃1min, 35个循环:72℃10min,4℃保存。PCR产物进行琼脂糖凝胶电泳检测,用DNA凝胶回收试剂盒对目的 片段进行回收、纯化。回收产物用T-DNA连接酶连接到pMD18-T-simple载体,连接产物转化大肠杆菌 DH5α(由本实验室保存),涂于氨苄青霉素抗性的LB平板37℃暗培养12h,挑取阳性单克隆送到南京金 斯瑞生物科技有限公司测序分析
第 2 期 张飞雪ꎬ等:不结球白菜醛酮还原酶 BcAKR4C9 基因的克隆及表达分析 在植物生长发育的各个阶段ꎬ植物会不可避免地遇到一些生物和非生物胁迫[1] ꎮ 研究表明:植物在 遭遇非生物胁迫时ꎬ活性氧与活性乙醛水平增加ꎬ导致植物的胁迫诱导损伤[2] ꎬ为了清除植物体内产生的 活性氧、活性乙醛等有毒物质ꎬ植物会利用大量具有氧化还原活性的酶类使有毒物质分解为毒性较小的乙 醇和碳水化合物[3] ꎬ而醛酮还原酶(AKR) 家族通常催化醛、酮、类固醇以及其他羰基化合物的还原[3] ꎮ AKR 家族在逆境防御中起重要作用ꎬ能够被多种逆境胁迫诱导[4-7] ꎻ在植物中它们起着反应性醛解毒、渗 透压生成和次级代谢物合成的关键作用[8] ꎮ AKR 家族ꎬ是以 NADP(H)为辅酶而催化一系列醛酮底物发生氧化还原反应ꎬ具有经典的(α / β)8 桶 状折叠结构ꎬ在进化上高度保守ꎮ 其中ꎬ(α / β)8 桶状结构羧基端的 3 个环(LooP A、LooP B、LooP C)的不 同组成和大小决定了其底物的特异性[9] ꎮ AKR 催化活性位点是由 N 末端(Asp50、Tyr55、Lys84) 所构 成[9] ꎬ催化四联体(Asp50、Tyr55、Lys84、His117)被认为是醛酮还原酶负责底物催化的关键氨基酸[10] ꎻ另 外ꎬ发现 C 末端形成的高度保守的氨基酸残基(Ser271、Arg276)在辅因子结合中起重要作用[9] ꎮ AKR4C 亚家族与植物的耐受性相关[3] ꎮ AKR4C 能够被高 CO2 浓度和高光照下诱导表达ꎬ使细胞内 光合速率增强[11] ꎮ 在单子叶植物中首先鉴定到的 AKR4C1 被认为是耐旱性相关的蛋白并能够被脱落酸 诱导表达[12] ꎮ AKR4C9 为 AKR4C 亚家族的成员ꎬ在逆境胁迫中能够起到醛酮解毒的作用[13] ꎬ被认为是保 护植物免受应激氧化胁迫的重要的候选基因[3] ꎮ 在大麦中过表达拟南芥 AKR4C9 基因可以在氧化胁迫和 镉胁迫中增强其醛酮解毒反应ꎬ转基因植株呼吸速率降低ꎬ叶绿素含量增加并且光合速率提高[13] ꎻ山梨醇 浓度相对于野生型提高 2~4 倍ꎬ其耐寒性和再生能力得到显著提高[14] ꎮ 本研究以不结球白菜品种‘苏州青’为试验材料ꎬ克隆了 AKR4C9 同源基因ꎬ并且进行了相关的生物 信息学分析ꎮ 利用实时荧光定量 PCR 技术分别测定了高温、低温、NaCl、伤害处理下的基因表达水平ꎬ以 及在脱落酸、茉莉酸甲酯和水杨酸激素处理下基因的表达变化ꎬ旨在进一步了解 BcAKR4C9 基因在不结球 白菜中的功能ꎮ 1 材料与方法 1.1 材料及处理 供试材料为不结球白菜(Brassica campestris ssp. chinensis Makino)品种‘苏州青’ꎬ由南京农业大学园艺学 院不结球白菜课题组提供ꎮ 将‘苏州青’种植于南京农业大学人工气候室ꎬ在植株 4 叶 1 心期时ꎬ选取生长整 齐一致的幼苗作为试验材料ꎮ 以 42 ℃热激和 4 ℃低温处理幼苗ꎬ处理设备为 RXZ 智能型人工气候培养箱ꎬ 光照强度为 100 μmolm -2s -1 ꎬ处理后 0、4、6、8 和 24 h 取样ꎬ每处理 10 株ꎬ3 次重复ꎮ 在每片叶上打 3 个孔 (保留圆片不致脱离ꎬ孔径大小为 1 cm)作为伤害处理ꎬ未打孔的作为对照ꎬ分别在 0、4、8、12 和 24 h 后取样ꎬ 每处理 10 株ꎬ3 次重复ꎮ 分别以 5、10、15、20、25 gL -1的 NaCl 溶液喷洒叶片背面ꎬ以清水为对照ꎬ然后用透光 塑料罩罩住ꎬ处理 12 h 后取样ꎬ每处理 10 株ꎬ3 次重复ꎮ 选用 2 mmolL -1水杨酸(SA)、50 μmolL -1茉莉酸甲 酯(JA)和 50 μmolL -1脱落酸(ABA)溶液分别喷施 4 叶 1 心期的叶片ꎬ然后用透光塑料罩罩住ꎬ处理时间为 0、4、8、12 和 24 hꎬ每处理 10 株ꎬ3 次重复ꎮ 材料收集后液氮速冻ꎬ存放-80 ℃保存备用ꎮ 1.2 RNA 的提取与 cDNA 的合成 RNA 提取试剂盒购自天根生化科技有限公司ꎬ具体方法参照 RpPureNApre 植物总 RNA 提取试剂盒说明 书ꎮ 用‘苏州青’叶片的总 RNA 为模板ꎬ按照 PrimeScript RT 试剂盒(TaKaRa)说明书反转录合成 cDNAꎮ 1.3 不结球白菜 BcAKR4C9 基因的克隆 根据大白菜数据库( http: / / brassicadb. org / brad / searchGene. php) 的基因序列编号 Bra000018ꎬ利用 Primer Premier 6.0 软件设计 1 对引物ꎬ引物分别为 AKR4C9 ̄F:ATGGCTAACGCAATTAGATꎬAKR4C9 ̄R: TTATATCTCGCCATCCCATAAGTꎮ 以不结球白菜‘苏州青’叶片 cDNA 作为模板进行 PCR 反应ꎬ反应体系 按照 TaKaRa 公司的 rTaq 酶说明书进行ꎮ 反应程序为:95 ℃ 5 minꎻ95 ℃ 30 sꎬ58 ℃ 30 sꎬ72 ℃ 1 minꎬ 35 个循环ꎻ72 ℃ 10 minꎬ4 ℃保存ꎮ PCR 产物进行琼脂糖凝胶电泳检测ꎬ用 DNA 凝胶回收试剂盒对目的 片段进行回收、纯化ꎮ 回收产物用 T4  ̄DNA 连接酶连接到 pMD18 ̄T ̄simple 载体ꎬ连接产物转化大肠杆菌 DH5α(由本实验室保存)ꎬ涂于氨苄青霉素抗性的 LB 平板 37 ℃暗培养 12 hꎬ挑取阳性单克隆送到南京金 斯瑞生物科技有限公司测序分析ꎮ 241

242 南京农业大学学报 第41卷 1.4序列分析 用BioEdit软件查找不结球白菜BeAKR-4C9基因序列的开放阅读框(ORF)。不同物种AKRC49的氨 基酸序列从NCBI(http://www.ncbi.nih.gov/)GenBank数据库中搜索获得,同源性计算利用NCBI网站 BLAST程序。氨基酸编码的蛋白质相对分子质量、理论等电点、不稳定指数等通过在线软件Protparam tool(htp:/web.Expasy.org/protparam/)完成。疏水性预测采用ProtScale(htp:/weh.expasy..org/ protscale/),信号肽预测采用Signalp4.0(htp://www.cbs.dtu.dk/services/SignalP/),跨膜区域预测采用 TMHMM2.0(http:/www.cbs.dtu.dk/services/TMHMM/),蛋白质二级结构预测采用SOPMA(htp:/ pbil.ibcp.f/)等在线工具。蛋白质三级结构分析通过SWISS-MODEL(htps:/www.swissmodel..expasy.. og)完成。多重序列比对通过DNAMAN6.0进行,进化树构建用MEGA5.2软件s进行。 1.5实时荧光定量PCR 实时荧光定量PCR体系参照SYBR PrimeScript RT-PCR KitⅡ试剂盒(TaKaRa,Japan)说明书。内参 基因为Actin(登录号:AF111812),内参引物序列为Actin-F:GTTGCTATCCAGGCTGTTCT,Actin-R:AGCGT- GAGGAAGAGCATAAC;AKR4C9引物序列为qAKR4C9-F:GCCGCAGCTGTTAAGATCGGAT;qAKR4C9-R: GGTCATGATCCGTACACCAG。PCR程序采用两步法:95℃30s;95℃5s,60℃30s,40个循环;设置 65℃到95℃的熔解曲线。目标基因表达量的计算按照△4△C,法。相对表达量等于2r,即相对定量是 基于对照和处理之间,目标基因和内参基因表达量的比值。所得数据用Excl2010软件分析。 2结果与分析 2.1不结球白菜BeAKR4C9基因的克隆和序列分析 测序结果表明:不结球白菜BeAKR-4C9基因与拟南芥序列长度一致,序列相似度为88.7%,包含1个 长度为948bp的开放式阅读框(ORF),编码315个氨基酸,包含AKR家族共有的催化四联体(Asp47、 Ty52、Lys81、His114)(图1)。预测该基因编码蛋白质相对分子质量为35.126×103,理论等电点为6.61,不 稳定指数为39.81,属于不稳定蛋白。ProtScale亲/疏水性预测显示:该蛋白属于疏水性蛋白(图2),总平 1 ATGGCTAACGCAATTAGATTCTTIGAGCTCAACACTGGCGCTAAGATCCCGTCGGTGGGTTTGGGAACATGGCAA 1 M A N A I R FF E LN T G A K I P S V G L G T W Q 76 GCCICTCCGGGTCITGICGGTGATGCAGICGCCGCAGCTGTTAAGATCGGATATCGTCACATTGAIIGCGCICAG 26 A S P G L V G D A V AAA V K I G Y R H IDC A Q 151 ATCTACGGCAACGAAAAAGAGATTGGITCAGITTTGAAAAAATTGITTGAAGACAATGTAGTGAAACGGGAGGAG 51 IYG N E K E I G S V L KK L F E D N VV K R EE 226 TTGTTCATCACTTCCAAACTCTGGTGTACGGATCATGACCCTCAAGATGIGCCTGAGGCACTAAAAAGAACICTA 76 L F I T SKL W C T D H D P Q D V P E A L K R T L 301 CAGGATTTGCAGCTTGACTACGTCGATCTCTATTTGATACATTGGCCTGTACGGATGAAGAAAGGTTCTGTTGGA 101 Q D L Q L D Y V D L Y L IH W P V R M KK G S V G 376 GCTAAGCCCGAGAACCTTATGCCTGTAGATATTCCTAGCACATGGAAAGCGATGGAAGCACICTATGATICGGGC I26 A K PE N L M P V D⊥P S T W K A M E A L¥DSG 451 AAGGCACGGGCCATAGGTGTAAGCAATTTCICCACCAAGAAGCTAGCTACTCTCITGGAGITAGCTCGTGITCCT 151 K A R A I G V S N F S T KK L A T LL E L A R VP 526 CCTGCGGTTAACCAGGITGAATGICATCCTTCTIGGCAACAGACTAAGCTACGAGAGITCTGCAAAACCAAAGGA 176 P A V N Q V E C H P S W QQ T K L R E F C K T K G 601 GTTCACCTCACCGCATACTCTCCATTGGGTTCICCAGGAACAACGIGGCTCAAGAGCGATGITTIGAAGAACCCT 201 V H L T A Y S P L G S P G TT W L K S D V L K N P 676 ATACTGAACACTGTTGCTGAAAAACTAGGAAAGICICCIGCTCAAGTCGCACTTCGCTGGGGACTTCAAATGGGT 226 I L N T V A E K L G K S P A Q V A L R W G L Q M G 751 AACAGTGTGCTICCTAAAAGTACCAATGAAGGAAGAATTAGAGCGAACTTTGAGGTTTTTGACTGGTCAATACCC 251 N S V L P K S T N E G R I R A N F E V F DW S I P 826 GATGACTTGTTTGCCAAGTTTTCAGAGATTGAGGAGGCTAGGTTATTGAATGCTTCCTTCTTTGTTCATGAGACA 276 DD L F A K F S E I EE A R LL N A S FF V H E T 901 CTGAGCCCTTATAAGICICTTGAAGACTTATGGGATGGCGAGATATAA 301 L S P Y K S L E D L W D G E I* 图1不结球白菜BcAKR4C9基因的核苷酸序列及其编码的氨基酸序列 Fig.1 Nucleotide sequence and its encoded amino acid sequence of BcAKR4C9 gene from non-heading Chinese cabbage 方框内为催化四联体(Asp47、Tyr52、Lys81、Hisl14)位点。The box is a catalytic residue(Asp47,Tyr52, Lys81,and His114)locus
南 京 农 业 大 学 学 报 第 41 卷 1.4 序列分析 用 BioEdit 软件查找不结球白菜 BcAKR4C9 基因序列的开放阅读框(ORF)ꎮ 不同物种 AKRC49 的氨 基酸序列从 NCBI( http: / / www. ncbi. nih. gov / ) GenBank 数据库中搜索获得ꎬ同源性计算利用 NCBI 网站 BLAST 程序ꎮ 氨基酸编码的蛋白质相对分子质量、理论等电点、不稳定指数等通过在线软件 Protparam tool ( http: / / web. Expasy. org / protparam / ) 完 成ꎮ 疏 水 性 预 测 采 用 ProtScale ( http: / / web. expasy. org / protscale / )ꎬ信号肽预测采用 Signalp 4.0( http: / / www. cbs. dtu. dk / services/ SignalP / )ꎬ跨膜区域预测采用 TMHMM 2. 0 ( http: / / www. cbs. dtu. dk / services/ TMHMM/ )ꎬ蛋白质二级结构预测采用 SOPMA ( http: / / pbil.ibcp. fr/ ) 等在线工具ꎮ 蛋白质三级结构分析通过 SWISS ̄MODEL ( https: / / www.swissmodel. expasy. org / )完成ꎮ 多重序列比对通过 DNAMAN 6.0 进行ꎬ进化树构建用 MEGA 5.2 软件[15]进行ꎮ 1.5 实时荧光定量 PCR 实时荧光定量 PCR 体系参照 SYBR PrimeScript RT ̄PCR Kit Ⅱ试剂盒(TaKaRaꎬJapan)说明书ꎮ 内参 基因为 Actin(登录号:AF111812)ꎬ内参引物序列为 Actin ̄F:GTTGCTATCCAGGCTGTTCTꎬActin ̄R:AGCGT ̄ GAGGAAGAGCATAACꎻAKR4C9 引物序列为 qAKR4C9 ̄F:GCCGCAGCTGTTAAGATCGGATꎻ qAKR4C9 ̄R: GGTCATGATCCGTACACCAGꎮ PCR 程序采用两步法:95 ℃ 30 sꎻ95 ℃ 5 sꎬ60 ℃ 30 sꎬ40 个循环ꎻ设置 65 ℃到 95 ℃的熔解曲线ꎮ 目标基因表达量的计算按照 ΔΔCT 法ꎮ 相对表达量等于 2 -ΔΔCT ꎬ即相对定量是 基于对照和处理之间ꎬ目标基因和内参基因表达量的比值ꎮ 所得数据用 Excel 2010 软件分析ꎮ 图 1 不结球白菜 BcAKR4C9 基因的核苷酸序列及其编码的氨基酸序列 Fig 1 Nucleotide sequence and its encoded amino acid sequence of BcAKR4C9 gene from non ̄heading Chinese cabbage 方框内为催化四联体(Asp47、Tyr52、Lys81、His114) 位点ꎮ The box is a catalytic residue( Asp47ꎬTyr52ꎬ Lys81ꎬand His114)locus. 2 结果与分析 2.1 不结球白菜 BcAKR4C9 基因的克隆和序列分析 测序结果表明:不结球白菜 BcAKR4C9 基因与拟南芥序列长度一致ꎬ序列相似度为 88.7%ꎬ包含 1 个 长度为 948 bp 的开放式阅读框(ORF)ꎬ编码 315 个氨基酸ꎬ包含 AKR 家族共有的催化四联体(Asp47、 Tyr52、Lys81、His114)(图 1)ꎮ 预测该基因编码蛋白质相对分子质量为 35.126×10 3 ꎬ理论等电点为 6.61ꎬ不 稳定指数为 39.81ꎬ属于不稳定蛋白ꎮ ProtScale 亲/ 疏水性预测显示:该蛋白属于疏水性蛋白(图 2)ꎬ总平 242
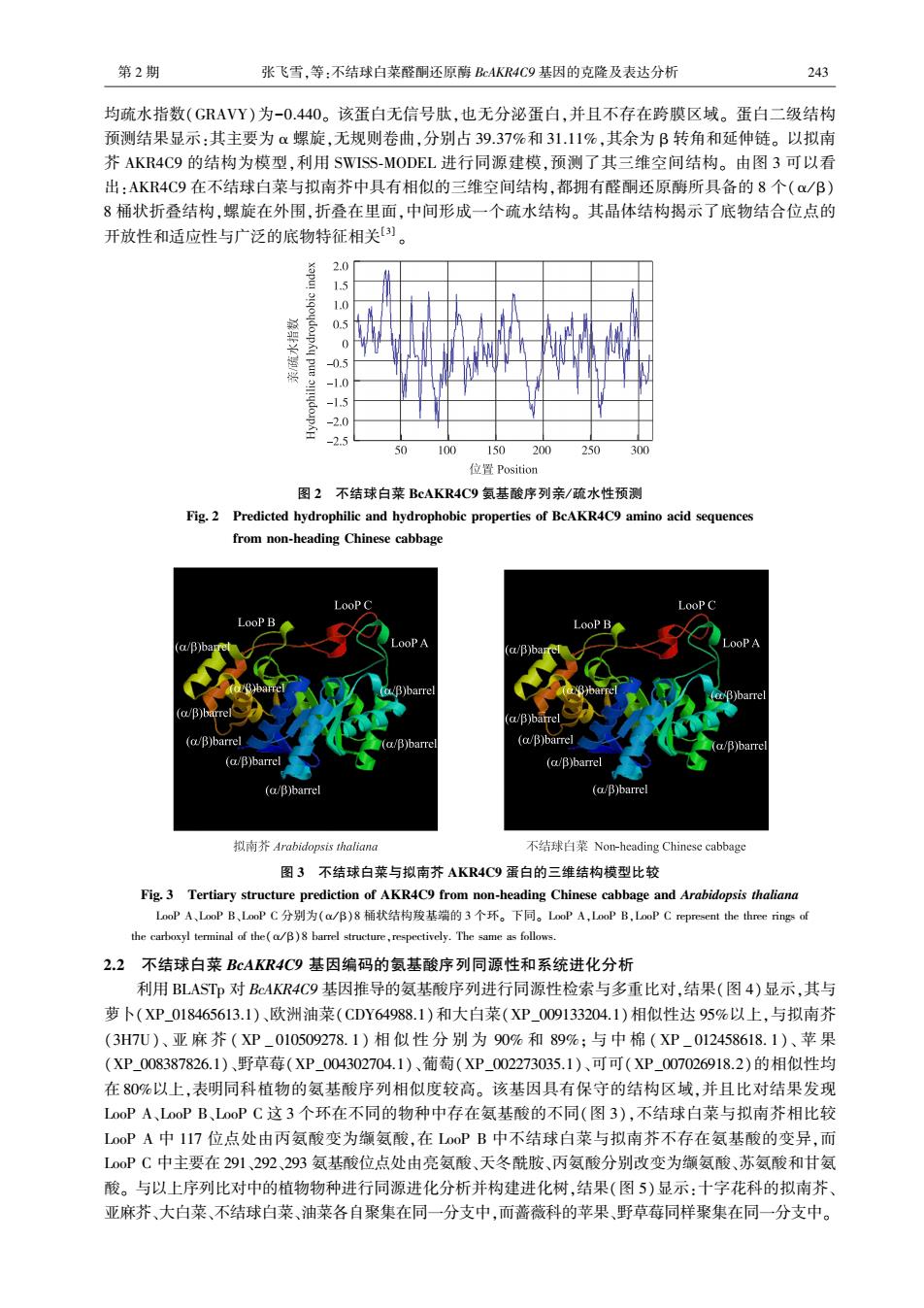
第2期 张飞雪,等:不结球白菜醛酮还原酶BcAKR4C9基因的克隆及表达分析 243 均疏水指数(GAVY)为-0.440。该蛋白无信号肽,也无分泌蛋白,并且不存在跨膜区域。蛋白二级结构 预测结果显示:其主要为α螺旋,无规则卷曲,分别占39.37%和31.11%,其余为B转角和延伸链。以拟南 芥AKR4C9的结构为模型,利用SWISS-MODEL进行同源建模,预测了其三维空间结构。由图3可以看 出:AKR4C9在不结球白菜与拟南芥中具有相似的三维空间结构,都拥有醛酮还原酶所具备的8个(α/B) 8桶状折叠结构,螺旋在外围,折叠在里面,中间形成一个疏水结构。其晶体结构揭示了底物结合位点的 开放性和适应性与广泛的底物特征相关[) 2.0 1.5 1.0 0.5 0.5 -1.0 -1.5 -2.0 -2.5 50 100150200250 300 位置Position 图2不结球白菜BcAKR4C9氨基酸序列亲/疏水性预测 Fig.2 Predicted hydrophilic and hydrophobic properties of BeAKR4C9 amino acid sequences from non-heading Chinese cabbage LooP C LooP C LooP B LooP B a/净 /B)b8 ooP A (o/B)barre (a/B)barre (a/B)barrel a/B)barre (a/B)barrel (a/β)barrel (a/B)barrel (a/B)barrel (aβ)barrel 拟南芥Arabidopsis thaliana 不结球白菜Nom-heading Chinese cabbage 图3不结球白莱与拟南芥AKR4C9蛋白的三维结构模型比较 Fig.3 Tertiary structure prediction of AKR4C9 from non-heading Chinese cabbage and Arabidopsis thaliana LooP A、LooP B、LooP C分别为(a/B)8桶状结构羧基端的3个环。下同。LooP A,LooP B,LooP C represent the three rings of the carboxyl terminal of the(a/B)8 barrel structure,respectively.The same as follows. 2.2不结球白菜BAKR4C9基因编码的氨基酸序列同源性和系统进化分析 利用BLASTp对BeAKR4C9基因推导的氨基酸序列进行同源性检索与多重比对,结果(图4)显示,其与 萝卜(XP_018465613.1)、欧洲油菜(CDY64988.1)和大白菜(XP_009133204.1)相似性达95%以上,与拟南芥 (3H7U)、亚麻芥(XP_010509278.1)相似性分别为90%和89%;与中棉(XP_012458618.1)、苹果 (XP008387826.1)、野草莓(XP_004302704.1)、葡萄(XP002273035.1)、可可(XP007026918.2)的相似性均 在80%以上,表明同科植物的氨基酸序列相似度较高。该基因具有保守的结构区域,并且比对结果发现 LooP A、LooP B、LooP C这3个环在不同的物种中存在氨基酸的不同(图3),不结球白菜与拟南芥相比较 LooP A中117位点处由丙氨酸变为缬氨酸,在LooP B中不结球白菜与拟南芥不存在氨基酸的变异,而 L00PC中主要在291、292、293氨基酸位点处由亮氨酸、天冬酰胺、丙氨酸分别改变为缬氨酸、苏氨酸和甘氨 酸。与以上序列比对中的植物物种进行同源进化分析并构建进化树,结果(图5)显示:十字花科的拟南芥、 亚麻芥、大白菜、不结球白菜、油菜各自聚集在同一分支中,而蔷薇科的苹果、野草莓同样聚集在同一分支中
第 2 期 张飞雪ꎬ等:不结球白菜醛酮还原酶 BcAKR4C9 基因的克隆及表达分析 均疏水指数(GRAVY)为-0.440ꎮ 该蛋白无信号肽ꎬ也无分泌蛋白ꎬ并且不存在跨膜区域ꎮ 蛋白二级结构 预测结果显示:其主要为 α 螺旋ꎬ无规则卷曲ꎬ分别占 39.37%和 31.11%ꎬ其余为 β 转角和延伸链ꎮ 以拟南 芥 AKR4C9 的结构为模型ꎬ利用 SWISS ̄MODEL 进行同源建模ꎬ预测了其三维空间结构ꎮ 由图 3 可以看 出:AKR4C9 在不结球白菜与拟南芥中具有相似的三维空间结构ꎬ都拥有醛酮还原酶所具备的 8 个(α / β) 8 桶状折叠结构ꎬ螺旋在外围ꎬ折叠在里面ꎬ中间形成一个疏水结构ꎮ 其晶体结构揭示了底物结合位点的 开放性和适应性与广泛的底物特征相关[3] ꎮ 图 2 不结球白菜 BcAKR4C9 氨基酸序列亲/ 疏水性预测 Fig 2 Predicted hydrophilic and hydrophobic properties of BcAKR4C9 amino acid sequences from non ̄heading Chinese cabbage 图 3 不结球白菜与拟南芥 AKR4C9 蛋白的三维结构模型比较 Fig 3 Tertiary structure prediction of AKR4C9 from non ̄heading Chinese cabbage and Arabidopsis thaliana LooP A、LooP B、LooP C 分别为(α/ β)8 桶状结构羧基端的 3 个环ꎮ 下同ꎮ LooP AꎬLooP BꎬLooP C represent the three rings of the carboxyl terminal of the(α/ β)8 barrel structureꎬrespectively. The same as follows. 2.2 不结球白菜 BcAKR4C9 基因编码的氨基酸序列同源性和系统进化分析 利用 BLASTp 对 BcAKR4C9 基因推导的氨基酸序列进行同源性检索与多重比对ꎬ结果(图 4)显示ꎬ其与 萝卜(XP_018465613.1)、欧洲油菜(CDY64988.1)和大白菜(XP_009133204.1)相似性达 95%以上ꎬ与拟南芥 (3H7U)、 亚 麻 芥 ( XP _ 010509278. 1) 相 似 性 分 别 为 90% 和 89%ꎻ 与 中 棉 ( XP _ 012458618. 1)、 苹 果 (XP_008387826.1)、野草莓(XP_004302704.1)、葡萄(XP_002273035.1)、可可(XP_007026918.2)的相似性均 在 80%以上ꎬ表明同科植物的氨基酸序列相似度较高ꎮ 该基因具有保守的结构区域ꎬ并且比对结果发现 LooP A、LooP B、LooP C 这 3 个环在不同的物种中存在氨基酸的不同(图 3)ꎬ不结球白菜与拟南芥相比较 LooP A 中 117 位点处由丙氨酸变为缬氨酸ꎬ在 LooP B 中不结球白菜与拟南芥不存在氨基酸的变异ꎬ而 LooP C 中主要在 291、292、293 氨基酸位点处由亮氨酸、天冬酰胺、丙氨酸分别改变为缬氨酸、苏氨酸和甘氨 酸ꎮ 与以上序列比对中的植物物种进行同源进化分析并构建进化树ꎬ结果(图 5)显示:十字花科的拟南芥、 亚麻芥、大白菜、不结球白菜、油菜各自聚集在同一分支中ꎬ而蔷薇科的苹果、野草莓同样聚集在同一分支中ꎮ 243
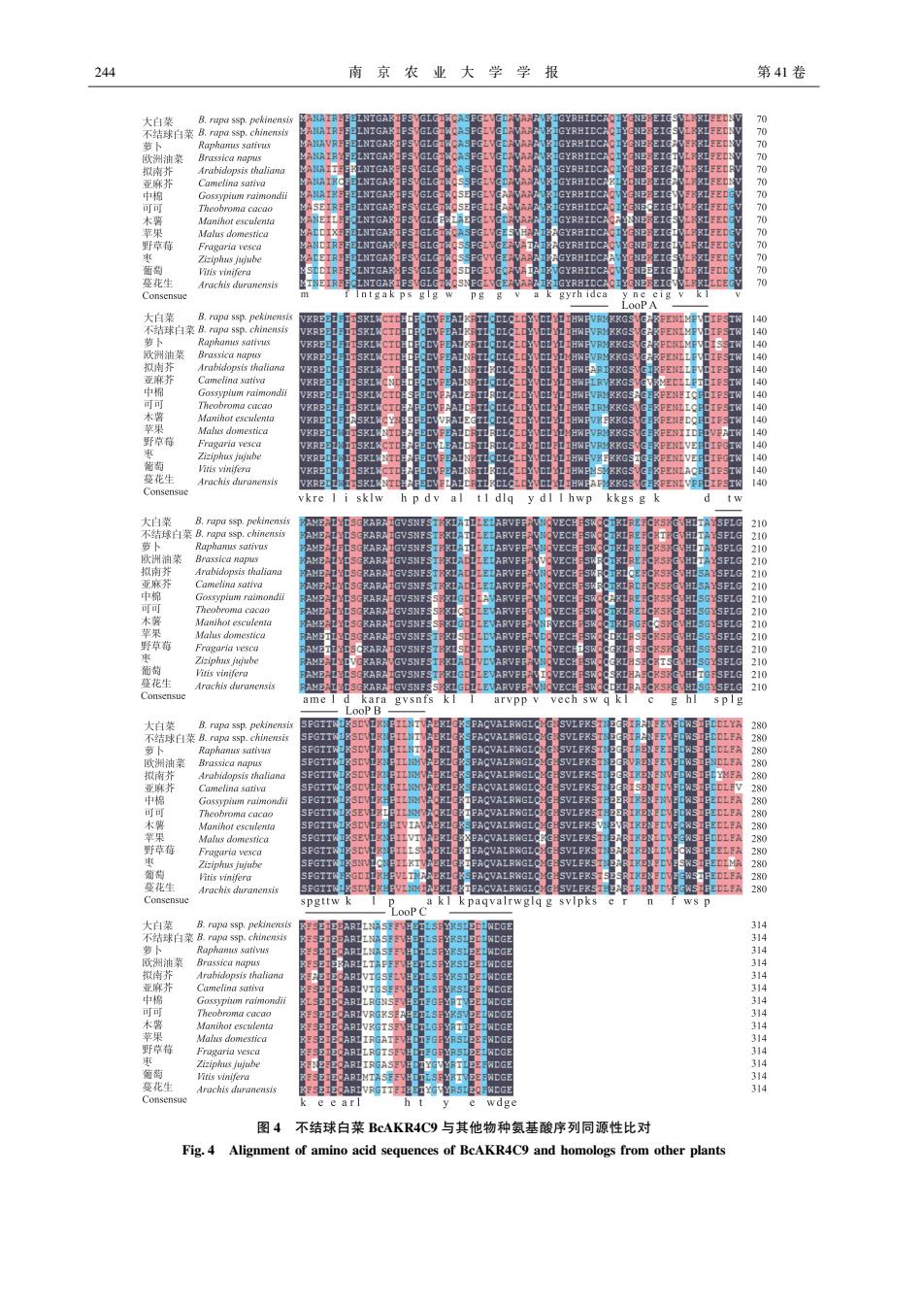
244 南京农业大学学报 第41卷 大白克 B.rapa ssp.pekinensis 不结球白菜B.rapa ssp.chinensi的 萝下 欧洲油菜 BrG3CRR见S 南芥 Arabidopsis thaliana 麻护 Camelina sativa 中 Gossypium raimondii 可 Theobroma cacao Manihot esculente Malus domestica 野草莓 Fraparia vesca GL 0000000000000 Visis vinifera 蔓花生 Arachis duranensis Consensue n t g a l 00P 大白菜 RE 不结球白菜B,rapa ssp,chinensis 萝下 欧洲油菜 行a50样ng VKR 型福 Arabidopsis thali 亚麻芥 中棉 r0s5D/所aD7d月 VKRI 可 Theobroma cacao Maniho Maluts domestica 野草莓 Frogaria vesca 4444444444 葡萄 Ziziphus jujub yitis vinijera 蔓花生 Arachis durane VKRE 40 Consensue vkre sklw dy kkgs t w 大白菜 rapa ssp.pekinen 210 不结球白菜B.rapa ssp.chinensis 萝卜 欧洲油菜 Brassica napu 以南 Arabidopsis thallang 亚底 中 木薯 Manihot esculenta 苹果 Malus domestic Fraearia vesca 葡韵 itis vin Arachis duranensis AME Conse ame gvsnfs arvpp vech 大白菜 B rana ssp.peslnensis 不结球白菜B.rapa ssp.chinensi Raphanus sativus 次用油 Brassica napus Arabidopsis thaliano Camelina sativa 中棉 G02时add 可可 Theobroma cacao 尊 苹用 Malts domestica 野草 Ziziphus jujub Vitis vinifera 蔓花生 280 Consensue spgttw ws p 大白菜 B.rapa ssp.pekinenst e! 不结球白菜B.apa ssp.chinens 34 sica nap Arabidoosis thaliana Camelina sativa 中棉 Gossypium raimo 可可 Theobroma cacao Manihot esculenta 苹果 Malus domestic 野草 Ziziphus tujube 葡萄 vinifera 314 蔓花生 Arachis duranensis 314 Consensue eearl e wdge 图4不结球白菜BeAKR4C9与其他物种氨基酸序列同源性比对 Fig.4 Alignment of amino acid sequences of BcAKR4C9 and homologs from other plants
南 京 农 业 大 学 学 报 第 41 卷 图 4 不结球白菜 BcAKR4C9 与其他物种氨基酸序列同源性比对 Fig 4 Alignment of amino acid sequences of BcAKR4C9 and homologs from other plants 244