
SV40早期启动子 组蛋白H2B CAAT ☐GC ■TATA
SV40 早期启动子 组蛋白H2B CAAT GC TATA
启动子与转录起始 1.启动子区的基本结构 ·启动子是一段位于结构基因5'端上游区的DNA序列,能活 化RNA聚合酶,使之与模板DNA准确地结合,并具有转录 起始的特异性。 Consensus sequences for Prokaryotic RNA polymerase binding promoter Transcription start site 35 -10 TTGACA TATAAT -35 region Pribnow box Structural DNA template genes (coding)strand RNA polymerase Eukaryotic promoter -75 -25 GGXCAATCT TATAAA CAAT box TATA box DNA template (coding)strand (x=any base) 2024/7/22 27
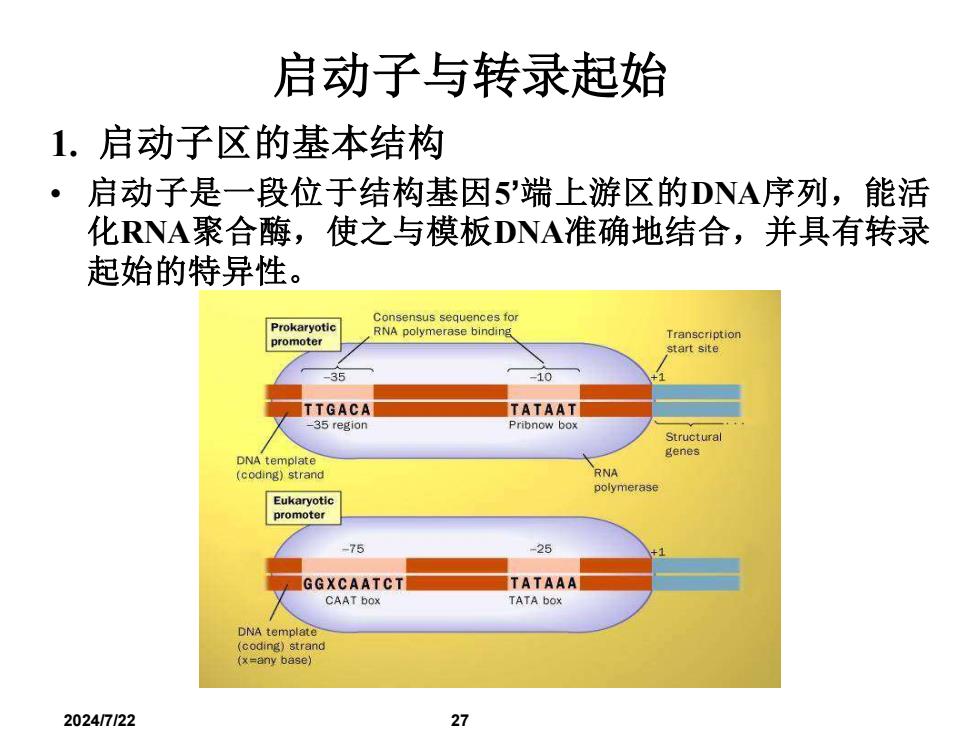
启动子与转录起始 1. 启动子区的基本结构 • 启动子是一段位于结构基因5’端上游区的DNA序列,能活 化RNA聚合酶,使之与模板DNA准确地结合,并具有转录 起始的特异性。 2024/7/22 27

1.启动子区的基本结构 ·原核生物中经对启动子的比较,它们有共有序列: -在上游10bp处为TATAAT,又称为Pribnow盒 -在上游35bp处为TTGACA。 -T82T84G78A65C54A45··T80Ag5T4sA60A50T96 Consensus sequences for RNA polymerase binding. Transcription start site -35 -10 +1 TTGACA 15176as0写 TATAAT 59 bases -35 region Pribnow box Coding sequences RNA polymerase 2024722 28
1. 启动子区的基本结构 • 原核生物中经对启动子的比较,它们有共有序列: – 在上游10bp处为TATAAT,又称为Pribnow盒 – 在上游35bp处为TTGACA。 – T82T84G78A65C54A45···T80A95T45A60A50T96 2024/7/22 28

。真核生物中: -TATA序列(TATA b0x):位于-25~35,使转录精确地起始, TATA序列也叫核心启动元件。 CCAAT序列(CAAT box):位于-70~-80,CAAT区主要控制转录 起始频率。 -GCCACACCC或GGGCGGG序列(GC box):位于-110~-80, 主要控制转录起始频率。 并非每个基因中都包含这3个区域。 2024/722 29
• 真核生物中: – TATA序列(TATA box):位于-25~-35,使转录精确地起始, TATA序列也叫核心启动元件。 – CCAAT序列(CAAT box) :位于-70~-80,CAAT区主要控制转录 起始频率。 – GCCACACCC或GGGCGGG序列(GC box):位于-110 ~-80, 主要控制转录起始频率。 ❖并非每个基因中都包含这3个区域。 2024/7/22 29

2.启动子区的识别 ·RNA聚合酶并不直接识别碱基对本身,而是通过氢键互补的方 式加以识别。 ·在启动子区DNA双螺旋结构中,存在特定方位的氢键供体和受 体(碱基上的基团),能与酶分子内也处于特定空间构象的氢 键受体与供体结合,从而相互识别。 3.RNA聚合酶与启动子区的结合 。 在识别阶段,聚合酶与启动子可逆性结合形成封闭二元复合物。 接着,结合处DNA双链解开,封闭复合物变为开放二元复合物。 20241722 30
2. 启动子区的识别 • RNA聚合酶并不直接识别碱基对本身,而是通过氢键互补的方 式加以识别。 • 在启动子区DNA双螺旋结构中,存在特定方位的氢键供体和受 体(碱基上的基团),能与酶分子内也处于特定空间构象的氢 键受体与供体结合,从而相互识别。 3. RNA聚合酶与启动子区的结合 • 在识别阶段,聚合酶与启动子可逆性结合形成封闭二元复合物。 接着,结合处DNA双链解开,封闭复合物变为开放二元复合物。 2024/7/22 30