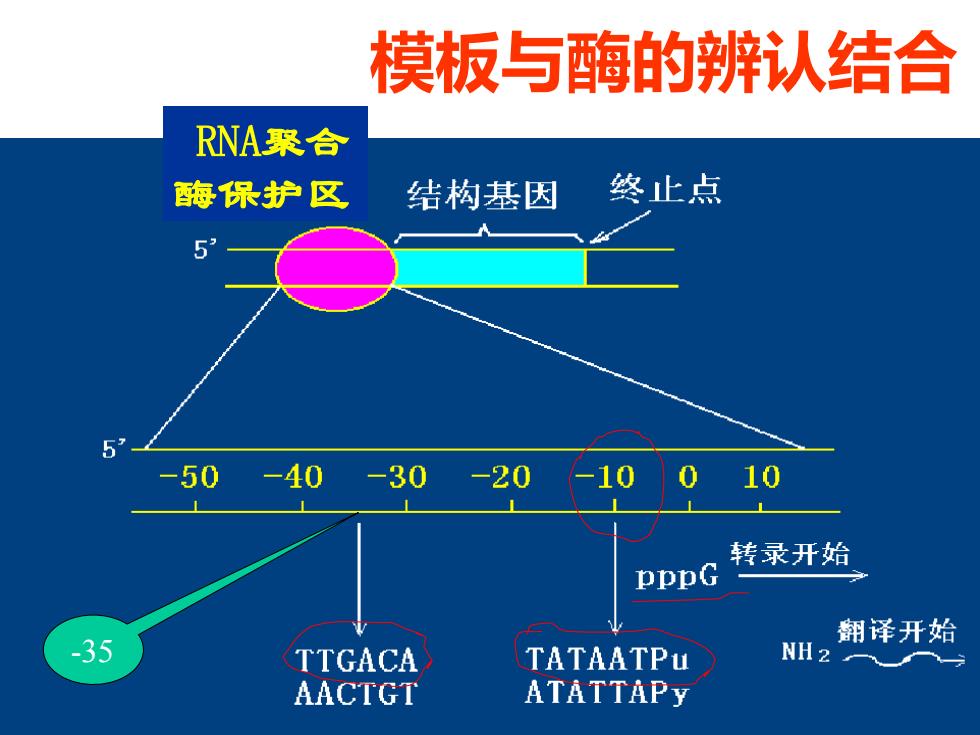
模板与酶的辨认结合 RNA聚合 酶保护区 结构基因 终止点 50-40-30-20 -10 0 10 转录开始 PPPG 翻译开始 35 TTGACA TATAATPu NH2 AACTGT ATATTAPY
模板与酶的辨认结合 RNA聚合 酶保护区 -35
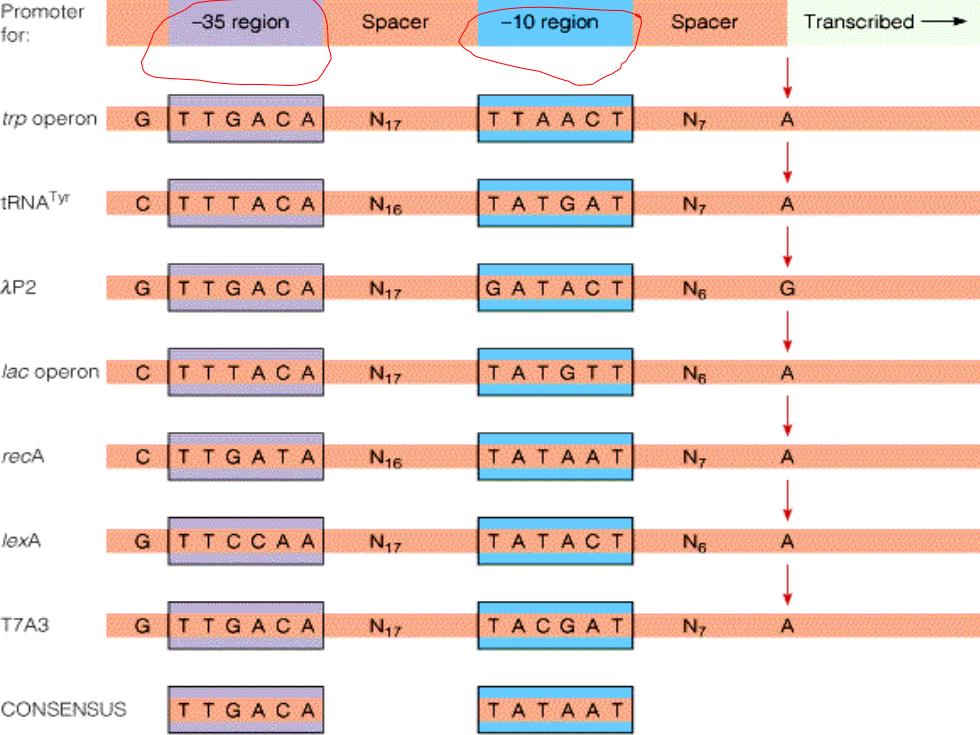
Promoter -35 region Spacer or: -10 region Spacer Transcribed- trp operon G TTGACA N17 N tRNATy TTTACA N16 TATGAT AP2 G GA T ACT G lac operon N17 recA TTGATA N16 lexA G N17 T7A3 G A T CONSENSUS TTGACA
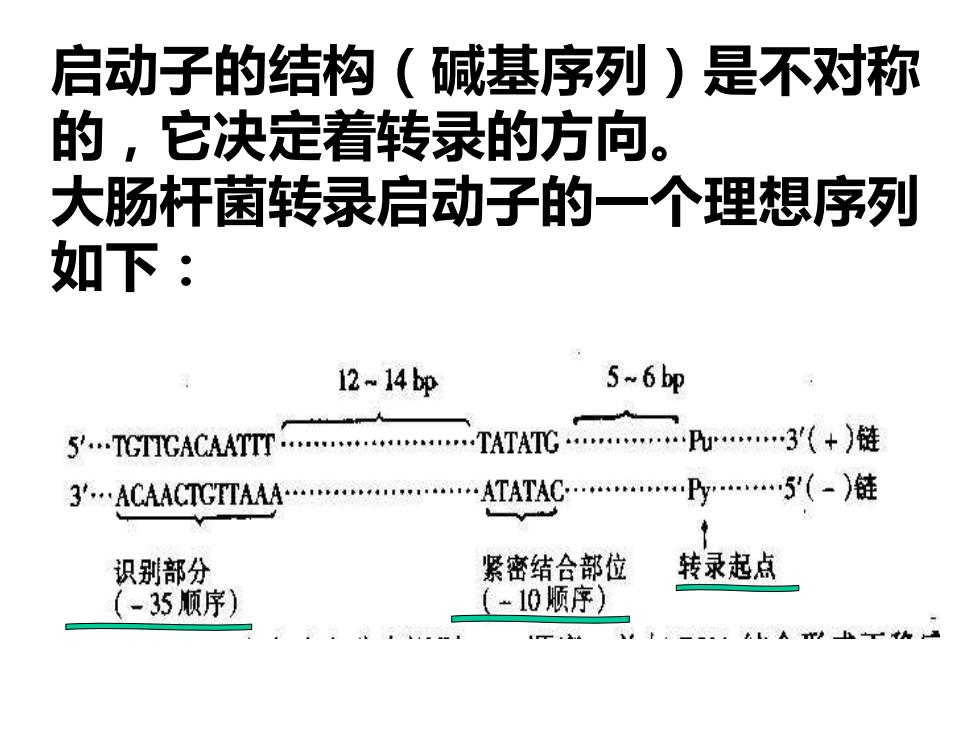
启动子的结构(碱基序列)是不对称 的,它决定着转录的方向。 大肠杆菌转录启动子的一个理想序列 如下: 12-14p 5~6bp 5.IGTIGACAATTT.TATATG.U3'(+)链 3.ACAACTGTTAAA.ATATACP.5(-)链 识别部分 紧密结合部位 转录起点 、-35顺序) (-10顺序) 上
启动子的结构(碱基序列)是不对称 的,它决定着转录的方向。 大肠杆菌转录启动子的一个理想序列 如下:

含有一条选单末端放射性 标记的同一DNA片段溶液 DNA 足迹法 用DNase处理使每 DNA 条链平均只有一个 酸切点 切点,被RM台 街结合的地方沿有 点 DNA片斯用放财性同位素 标记一会:用RNA聚合所保护 (结合)的DNA样品与不用此 薛保护的D州A样品分别用 DN50处里,控材条件使每个 DNA链只受一次切割:分肉标 记DNA片断并使之变性。用聚
DNA 足迹法 1 2

记DNA片断井使之变性。用聚 纯化刷处理DNA 丙烯酰胺餐胶电冰分开DNA片 片段并加以热变性 两,在X射线胶片上可以看到 放射标记区带,上面第一行为 ● 未切料DNA片断。失去的区带 说明RNA聚合的结合于DNA 聚丙烯酰胺散电泳分离 的地方,箭头示DNA迁移方向 通过足迹法研究启动子 DNA片段,放射自显影 生电泳图 末切DNA片段 DNA 迁移方向 RNA聚合酶结合 失去的区带,表期RNA 聚合每结台DNA的位点 保护的DNA顺序 为:-10区域和 三二 35区域。 图34足迹法探测蛋白质与DNA结合瓶序的示意图
• 通过足迹法研究启动子 3 4 RNA聚合酶结合 保护的DNA顺序 为: -10区域和 - 35区域