
表4-1国际互联网中的核酸序列限制性酶切分析资源 资源 网址 WebGene http://www bio.indiana.edu/-tjyin/WebGene/RE.html http://www.hyper-dimensions.com/person&tyin.html WebCutter2 http://www/ccsi.com/firstm 2025/5/27 BIOINFORMATICS 16
2025/5/27 BIOINFORMATICS 16 表4-1国际互联网中的核酸序列限制性酶切分析资源 资源 网址 WebGene http://www bio.indiana.edu/-tjyin/WebGene/RE.html http://www.hyper-dimensions.com/person&tyin.html WebCutter2 http://www/ccsi.com/firstm

同时,很多软件也能够识别REBASE限制 酶数据库。此处以使用BioEdit软件进行核酸 序列限制性酶切分析为例加以说明。将待分 析序列输入BioEdit软件的序列显示区后,按 照图4-3的说明即可完成分析过程,其结果见 图4-4。 数理与生物工程学院 2025/5/27 BIOINFORMATICS 17
2025/5/27 BIOINFORMATICS 数 理 与 生 物 工 程 学 院 17 同时,很多软件也能够识别REBASE限制 酶数据库。此处以使用BioEdit软件进行核酸 序列限制性酶切分析为例加以说明。将待分 析序列输入BioEdit软件的序列显示区后,按 照图4-3的说明即可完成分析过程,其结果见 图4-4
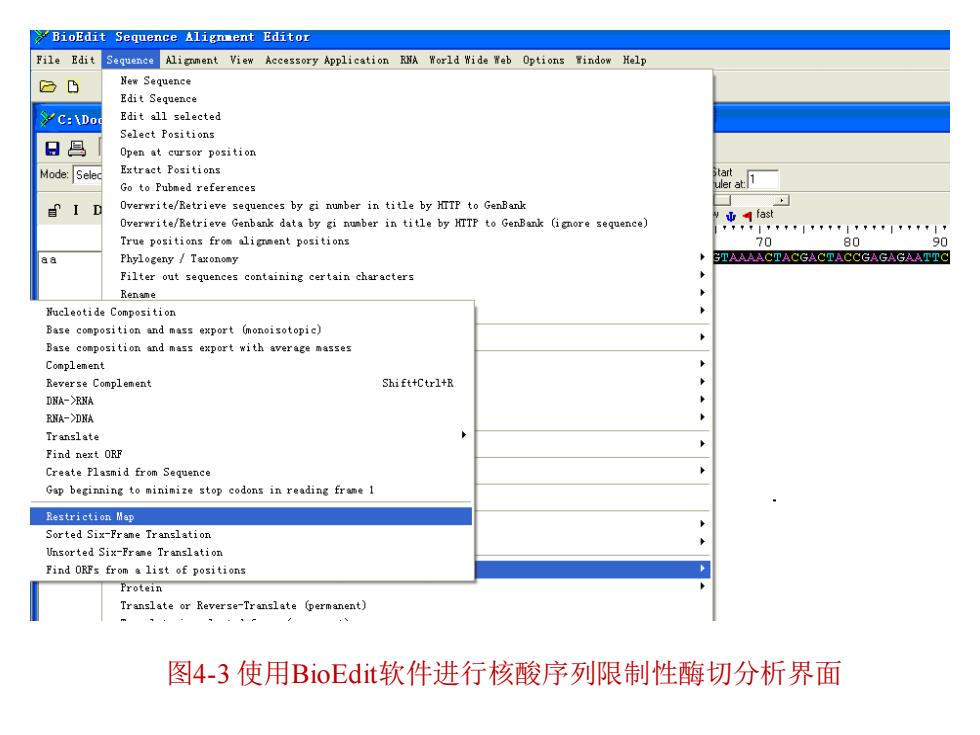
BioEdit Sequence Alignment Editor File Edit Socpono Aligment View Accessory Application RNA World Wide Web Options Window Help New Sequence Edit Sequence C:\Do Edit all selected Select Positions 日马 Open at cursor position Mode:Selec Extract Positions Go to Pubmed references I D Overwrite/Retrieve sequences by gi number in title by HITP to GenBank Overwrite/Retrieve Genbank data by gi nunber in title by HTTP to GenBank (ignore sequence) fast True positions from alignment positions 70 80 90 Phylogeny Taxonomy GTAAAACTACGACTACCGAGAGAATTC Filter out sequences containing certain characters Wucleotide Composition Base composition and mass export (monoisotopic) Base composition and mass export with average masses Complement Reverse Complement Shift+Ctrl+R DNA->RNA RNA-XDNA Translate Find next ORF Create Plasmid from Sequence Gap beginning to minimize stop codons in reading frame 1 Restriction Map Sorted Six-Frame Translation Unsorted Six-Frame Translation Find ORFs from a list of positions Protein Translate or Reverse-Translate (permanent) 图4-3使用BioEdit软件进行核酸序列限制性酶切分析界面
图4-3 使用BioEdit软件进行核酸序列限制性酶切分析界面
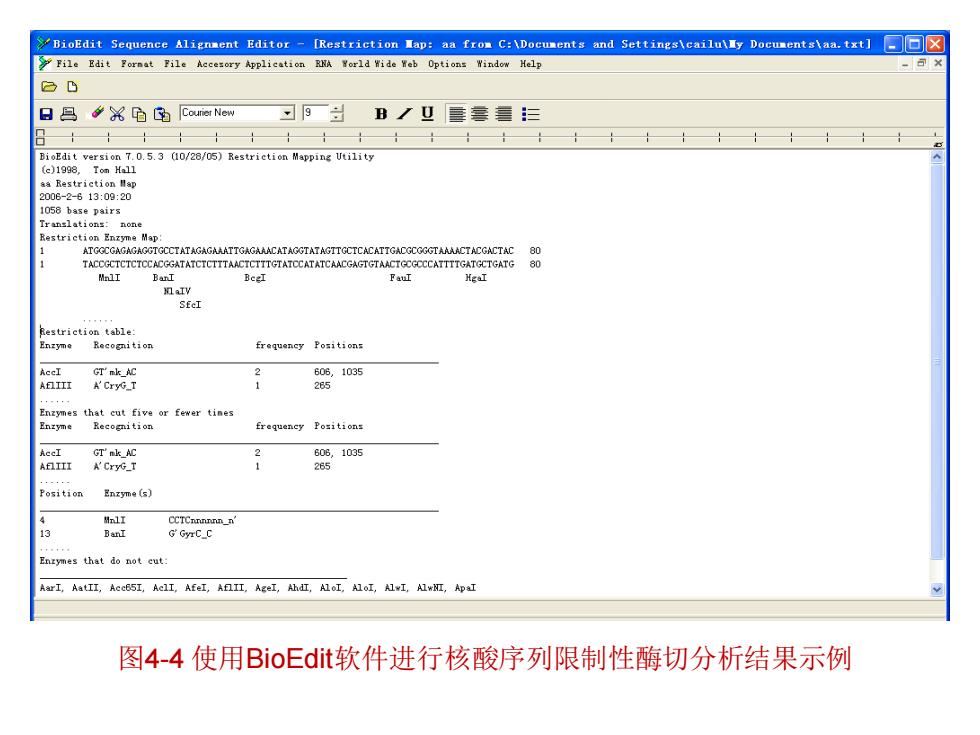
BioEdit Sequence Alignment Editor-[Restriction Nap:aa from C:\Documents and Settings\cailully Documents\aa.txt]x File Edit Format File Accesory Application RHA World Wide Web Options Window Help -× eD 日马XB南Courer New习9B/业套套复 BioEdit version 7.0.5.3 (10/28/05)Restriction Mapping Utility (c)1998,Ton Hall aa Restriction Hap 2006-2-613:09:20 1058 base pairs 88 BanI FauI MaTy SfeI Restriction table frequency Positions AccI GT'mk AC 606,1035 A'CryG_T 285 Enzymes that cut five or fewer tines Enzyme Recognition frequency Positions AGeT GT'nk AC 21 Position Enzyme (s) MnlI CCTCnnnnnn_n' 13 BanI GGyrC_C Enzymes that do not cut AarI,AatII,Ace5I,AclI,AfeI,AflII,AgeI,AhdI,AloI,AloI,AlwI,AlwNI,ApaI 图4-4使用BioEdit软件进行核酸序列限制性酶切分析结果示例
图4-4 使用BioEdit软件进行核酸序列限制性酶切分析结果示例

上述结果中给出了指定DNA序列的酶切 位点信息,为克隆鉴定和亚克隆提供了重要 信息。 数理与生物工程学院 2025/5/27 BIOINFORMATICS 20
2025/5/27 BIOINFORMATICS 数 理 与 生 物 工 程 学 院 20 上述结果中给出了指定DNA序列的酶切 位点信息,为克隆鉴定和亚克隆提供了重要 信息