
4.1.2.2分子质量、碱基组成、碱基分布 核酸序列的分子质量、碱基组成、碱基分布 等分析可通过一些常用软件如 BioEdit(http://www.mbio.ncsu.edu/BioEdit/bioedit.h tml),DNAMAN(http:/www.lynnon.com/)等直接 获得。将上述软件下载后即可进行此类分析。此 处以使用DNAMAN软件进行核酸序列(人环氧 数 化酶-2mRNA全序列,GenBank接受号AJ627251) 理 基本性质分析为例,可以看出,输出结果中 物 Composition(组成)和Percentage(百分比)一栏以 及Molecular Weight(分子质量)清楚地给出了关 程 于该条序列的有关结果。 院 2025/5/27 BIOINFORMATICS 11
2025/5/27 BIOINFORMATICS 数 理 与 生 物 工 程 学 院 11 4.1.2.2分子质量、碱基组成、碱基分布 核酸序列的分子质量、碱基组成、碱基分布 等 分 析 可 通 过 一 些 常 用 软 件 如 BioEdit(http://www.mbio.ncsu.edu/BioEdit/bioedit.h tml),DNAMAN(http://www.lynnon.com/)等直接 获得。将上述软件下载后即可进行此类分析。此 处以使用DNAMAN软件进行核酸序列(人环氧 化酶-2 mRNA全序列,GenBank接受号AJ627251) 基本性质分析为例,可以看出,输出结果中 Composition(组成)和Percentage(百分比)一栏以 及Molecular Weight(分子质量)清楚地给出了关 于该条序列的有关结果
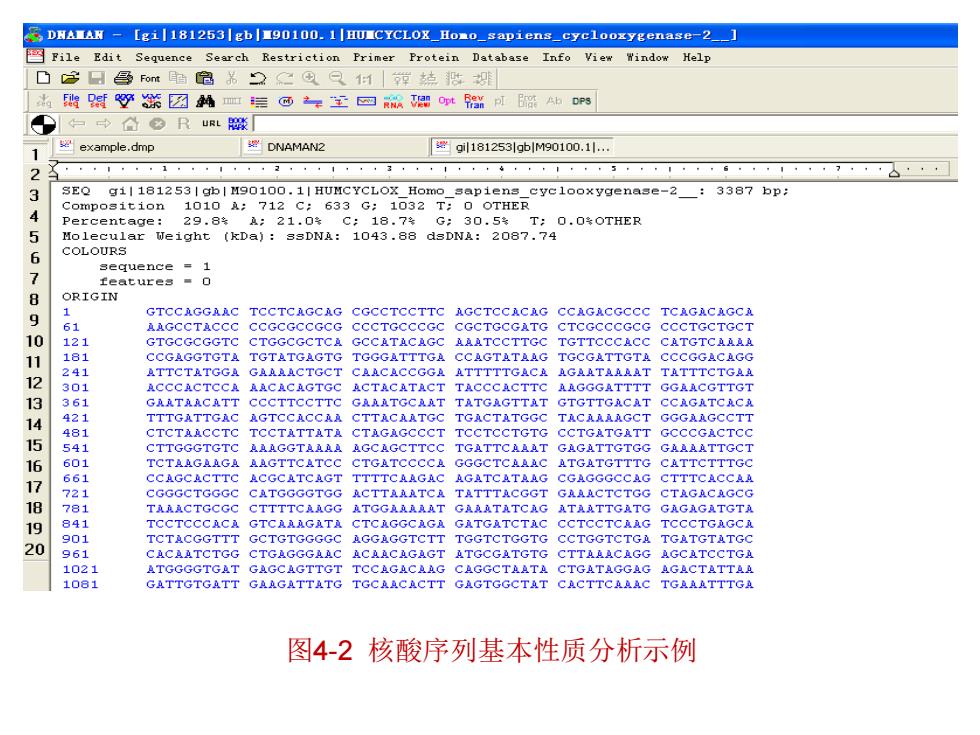
DNANAN [gil1812531gb|90100.1|HUHCYCLOX_Homo_sapiens_cyclooxygenase-2_] File Edit Sequence Search Restriction Primer Protein Database Info View Window Help ☐它可昌t宽二®反1萍绕概 =中合3RuL微 example.dmp DNAMAN2 gi1181253 lgblM90100.11., 2 3 SEQ gil181253gb|M90100.1|HUMCYCLOX Homo sapiens cyelooxygenase-2:3387 bp: 4 Composition 1010A:712C:633G:1032T:00THER Percentage: 29.8÷A:21.0÷C:18.7 G:30.5T:0.0%OTHER 56 Molecular Weight (kDa):SsDNA:1043.88 dsDNA:2087.74 COLOURS geC1enC世e=1 7 里色Q1上色号■口 ORIGIN 1 9 GTCCAGGAAC TCCTCAGCAG CGCCTCCTTC AGCTCCACAG CCAGACGCCC TCAGACAGCA 6 AAGCCTACCC CCGCGCCGCG CCCTGCCCGC CGCTGCGATG CTCGCCCGCG CCCTGCTGCT 10 121 GTGCGCGGTC CTGGCGCTCA GCCATACAGC AAATCCTTGC TGTTCCCACO CATGTCALAA 11 181 CCGAGGTGTA TGTATGAGTG TGGGATTTGA CCAGTATAAG TGCGATTGTA CCCGGACAGG 241 ATTCTATGGA GAAAACTGCT CAACACCGGA ATTTTTGACA TATTTCTGAA 12 301 ACCCACTCCA AACACAGTGC ACTACATACT TACCCACTTC AAGGGATTTT GGAACGTTGT 13 361 GAATAACATT CCCTTCCTTC GAAATGCAAT TATGAGTTAT GTGTTGACAT CCAGATCACA 14 421 TTTGATTGAC AGTCCACCAA TGACTATGG● TACAAAAGCT GGGAAGCCTT 481 CTCTAACCTC TCCTATTATA CTAGAGCCCT TCCTCCTGTG CCTGATGATT GCCCGACTCC 15 541 CTTGGGTGTC AGCAGCTTCC TGATTCAAAT GAGATTGTGG GAAAATTGCT 16 601 TCTAAGAAGA AAGTTCATCC CTGATCCCCA GGGCTCRAAC ATGATGTTTG CATTCTTTGC 17 661 CCAGCACTTC CGCATCAGT TTTTCAAGAC AGATCATAAG CGAGGGCCAG CTTTCACCAA 721 CGGGCTGGGC CATGGGGTGG ACTTAAATCA TATTTACGGT GAAACTCTGG CTAGACAGCG 8 781 TAAACTGCGC CTTTTCAAGG 熟TGGARAAAT GAARTATCAG ATAATTGATG GAGAGATGT熟 19 841 TCCTCCCACA GTCAAAGATA CTCAGGCAGA CCTCCTCAAG TCCCTGAGCA 901 TCTACGGTTT GCTGTGGGGC AGGAGGTCTT TGGTCTGGTG CCTGGTCTGA TGATGTATGC 20 961 CACAATCTGG CTGAGGGAAC ACAACAGAGT ATGCGATGTG CTTAAACAGG LGCATCCTG 1021 ATGGGGTGAT GAGCAGTTGT TCCAGACAAG CAGGCTAATA CTGATAGGAG AGACTATTAA 1081 GATTGTGATT GAAGATTATG TGCAACACTT GAGTGGCTAT CACTTCAAAC TGAAATTTGA 图4-2核酸序列基本性质分析示例
图4-2 核酸序列基本性质分析示例

4.1.2.3序列变换 进行序列分析时,经常需要对DNA序列进 行各种变换,例如反向序列、互补序列、互补 反向序列、显示DNA双链、转换为RNA序列等 。这些使用DNAMAN软件可以很容易地实现 ,这些功能集中在Sequence→Display,从中可 选择不同的序列变换方式对当前通道的序列进 行转换。 理与生物工程学院 2025/5/27 BIOINFORMATICS 13
2025/5/27 BIOINFORMATICS 数 理 与 生 物 工 程 学 院 13 4.1.2.3 序列变换 进行序列分析时,经常需要对DNA序列进 行各种变换,例如反向序列、互补序列、互补 反向序列、显示DNA双链、转换为RNA序列等 。这些使用DNAMAN软件可以很容易地实现 ,这些功能集中在Sequence→Display,从中可 选择不同的序列变换方式对当前通道的序列进 行转换
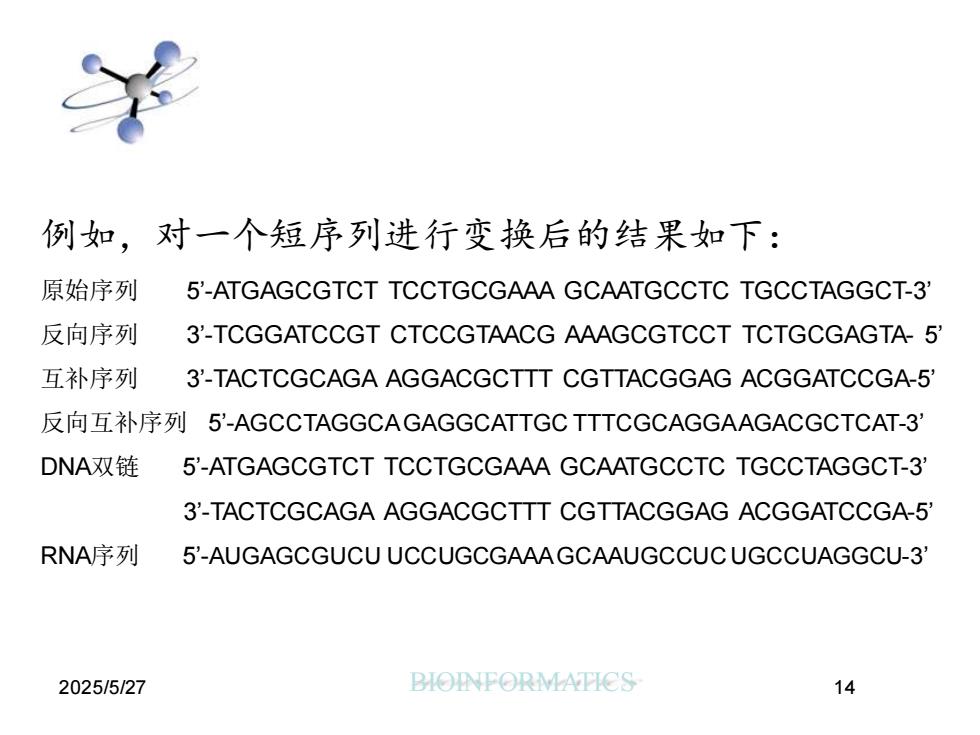
例如,对一个短序列进行变换后的结果如下: 原始序列 5-ATGAGCGTCT TCCTGCGAAA GCAATGCCTC TGCCTAGGCT-3 反向序列 3-TCGGATCCGT CTCCGTAACG AAAGCGTCCT TCTGCGAGTA-5 互补序列 3-TACTCGCAGA AGGACGCTTT CGTTACGGAG ACGGATCCGA-5 反向互补序列5'-AGCCTAGGCAGAGGCATTGC TTTCGCAGGAAGACGCTCAT-3' DNA双链 5-ATGAGCGTCT TCCTGCGAAA GCAATGCCTC TGCCTAGGCT-3 3'-TACTCGCAGA AGGACGCTTT CGTTACGGAG ACGGATCCGA-5 RNA序列 5-AUGAGCGUCU UCCUGCGAAAGCAAUGCCUCUGCCUAGGCU-3 2025/5/27 BIOINFORMATICS 14
2025/5/27 BIOINFORMATICS 14 例如,对一个短序列进行变换后的结果如下: 原始序列 5’-ATGAGCGTCT TCCTGCGAAA GCAATGCCTC TGCCTAGGCT-3’ 反向序列 3’-TCGGATCCGT CTCCGTAACG AAAGCGTCCT TCTGCGAGTA- 5’ 互补序列 3’-TACTCGCAGA AGGACGCTTT CGTTACGGAG ACGGATCCGA-5’ 反向互补序列 5’-AGCCTAGGCA GAGGCATTGC TTTCGCAGGA AGACGCTCAT-3’ DNA双链 5’-ATGAGCGTCT TCCTGCGAAA GCAATGCCTC TGCCTAGGCT-3’ 3’-TACTCGCAGA AGGACGCTTT CGTTACGGAG ACGGATCCGA-5’ RNA序列 5’-AUGAGCGUCU UCCUGCGAAA GCAAUGCCUC UGCCUAGGCU-3’

4.1.2.4限制性酶切分析 限制性酶切分析是分子生物学实验中的日常 工作之一。该方面最好的资源是限制酶数据库 (Restriction Enzyme Database REBASE) REBASE数据库中含有限制酶的所有信息,包括 甲基化酶、相应的微生物来源、识别序列位点、 裂解位,点、甲基化特异性、酶的商业来源以及公 开发表的和未发表的参考文献 理 (http://rebase.neb.com,http://www.neb.com/rebase) 物 。国际互联网上也有大量资源可供实时地进行限 程 制性酶切分析(表4-1)。 院 2025/5/27 BIOINFORMATICS 15
2025/5/27 BIOINFORMATICS 数 理 与 生 物 工 程 学 院 15 4.1.2.4 限制性酶切分析 限制性酶切分析是分子生物学实验中的日常 工作之一。该方面最好的资源是限制酶数据库 (Restriction Enzyme Database , REBASE) 。 REBASE数据库中含有限制酶的所有信息,包括 甲基化酶、相应的微生物来源、识别序列位点、 裂解位点、甲基化特异性、酶的商业来源以及公 开 发 表 的 和 未 发 表 的 参 考 文 献 (http://rebase.neb.com, http://www.neb.com/rebase) 。国际互联网上也有大量资源可供实时地进行限 制性酶切分析(表4-1)