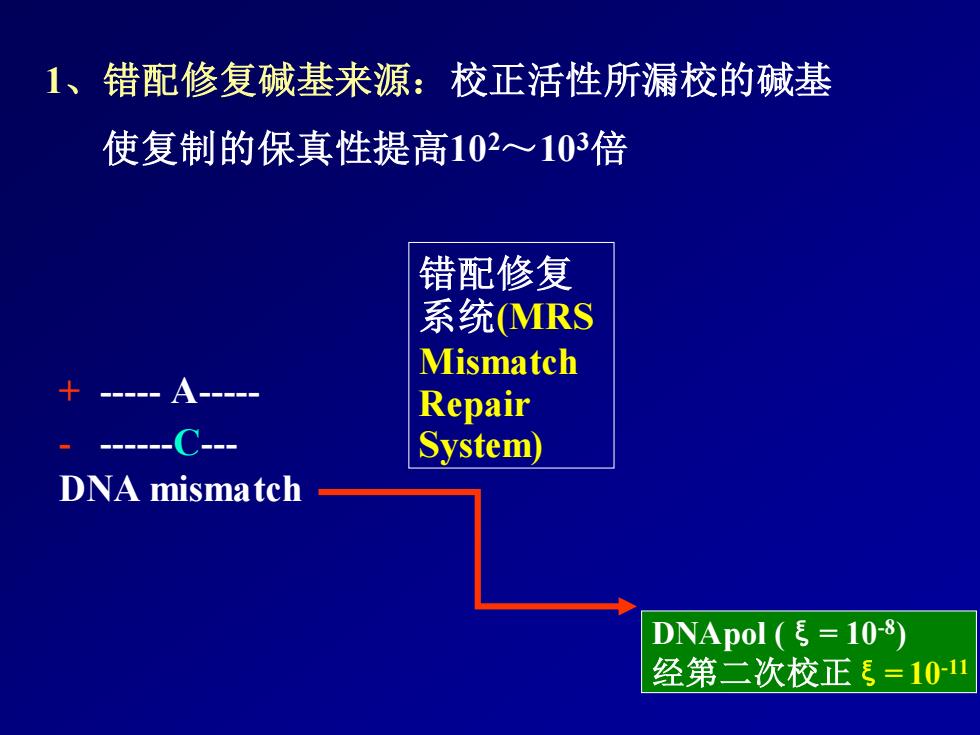
1、错配修复碱基来源:校正活性所漏校的碱基 使复制的保真性提高102~103倍 错配修复 系统MRS Mismatch Repair -C- System) DNA mismatch DNApol(ξ=10-8) 经第二次校正ξ=1011
DNA mismatch + - A- - -C- DNApol (ξ= 10-8 ) 经第二次校正ξ= 10-11 错配修复 系统(MRS Mismatch Repair System) 1、错配修复碱基来源:校正活性所漏校的碱基 使复制的保真性提高102~103倍

2、错配修复系统(Mismatch repair system) (1)组成 DNA腺嘌呤甲基化酶m6A甲基化酶) dam gene DNA polymerase Helicase SSB 外切核酸酶(I和Ⅶ) 连接酶 MCE (mismatch correct enzyme) 3 subunits mutH,L,S 识别新生链中非mA的GATC序列 扫描新生链中错配碱基 酶切含错配碱基的新生DNA区段
2、错配修复系统(Mismatch repair system) DNA polymerase Helicase SSB 外切核酸酶(Ⅰ和Ⅶ) 连接酶 MCE (mismatch correct enzyme) 3 subunits mutH, L, S DNA腺嘌呤甲基化酶(m dam gene 6A甲基化酶) 扫描新生链中错配碱基 识别新生链中非m6A 的GATC序列 酶切含错配碱基的新生DNA区段 (1)组成

CHs GATC DNA合成过程中的甲基化变化 CTAG CHs Replication DNA中的GATC(palindromic seq) CHs GATC 为m6A甲基化敏感位点 CTAG 平均每2kb左右有一GATC seq: For a short period following replication the template strand is CHs methylated and the new strand is not. 错配修复系统受甲基化的引导 5 CTAG Hemimethylated DNA GATC CTAG -5 CHg After a few minutes the new strand is Dam methylase methylated and the CHs two strands can no longer be distinguished. 5 GATC 3 CTAG -5 CHa C 工g CTAG
★ DNA合成过程中的甲基化变化 DNA中的GATC(palindromic seq.) 为m6A甲基化敏感位点 平均每2kb左右有一GATC seq. 错配修复系统受甲基化的引导
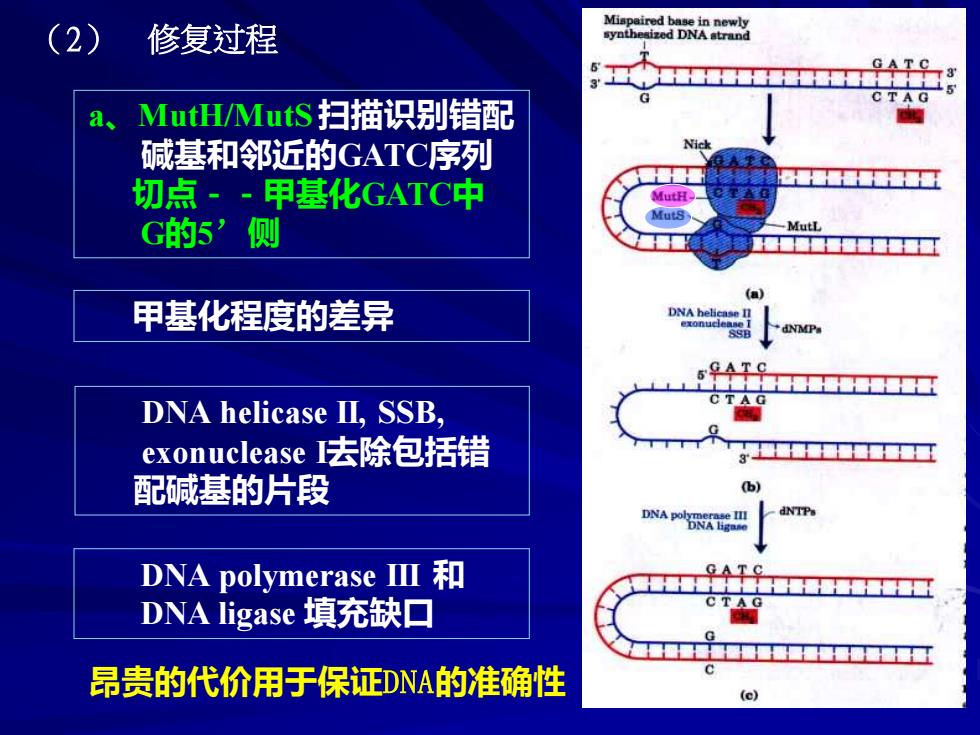
(2) 修复过程 GATC CTAG a、 MutH/MutS扫描识别错配 碱基和邻近的GATC序列 切点-甲基化GATC中 MutH Muts G的5’侧 MutL 甲基化程度的差异 DNA helieas dNMP ●TAC DNA helicase DⅡ,SSB, exonuclease去除包括错 配碱基的片段 (b) DNA dNTPs DNA polymeraseⅢ和 DNA ligase填充缺▣ CTAG 昂贵的代价用于保证DNA的准确性 (c)
甲基化程度的差异 a、MutH/MutS 扫描识别错配 碱基和邻近的GATC序列 切点--甲基化GATC中 G的5’侧 DNA helicase II, SSB, exonuclease I去除包括错 配碱基的片段 DNA polymerase III 和 DNA ligase 填充缺口 昂贵的代价用于保证DNA的准确性 (2) 修复过程
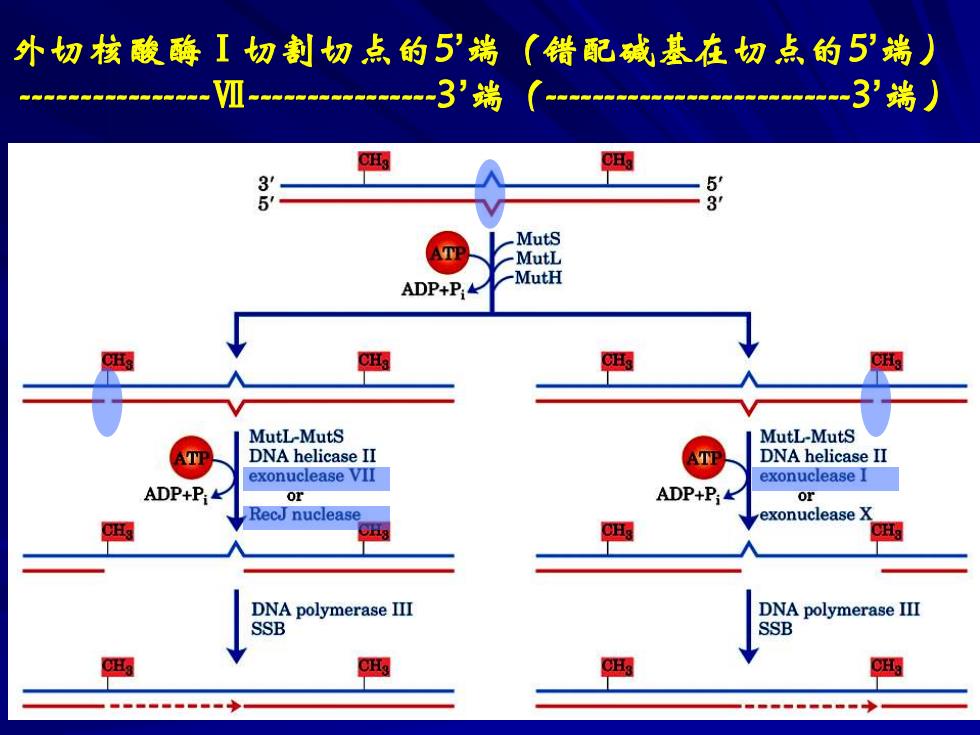
外切核酸酶I切割切点的5?端(错配碱基在切点的5'端) 3 5 MutS ATE MutL MutH ADP+P C CH MutL-MutS MutL-MutS 四 DNA helicase II DNA helicase II exonuclease VII exonuclease I ADP+P or ADP+P; RecJ nuclease exonuclease x H CHa DNA polymerase III DNA polymerase III SSB SSB
外切核酸酶Ⅰ切割切点的5’端(错配碱基在切点的5’端) -Ⅶ-3’端(-3’端)