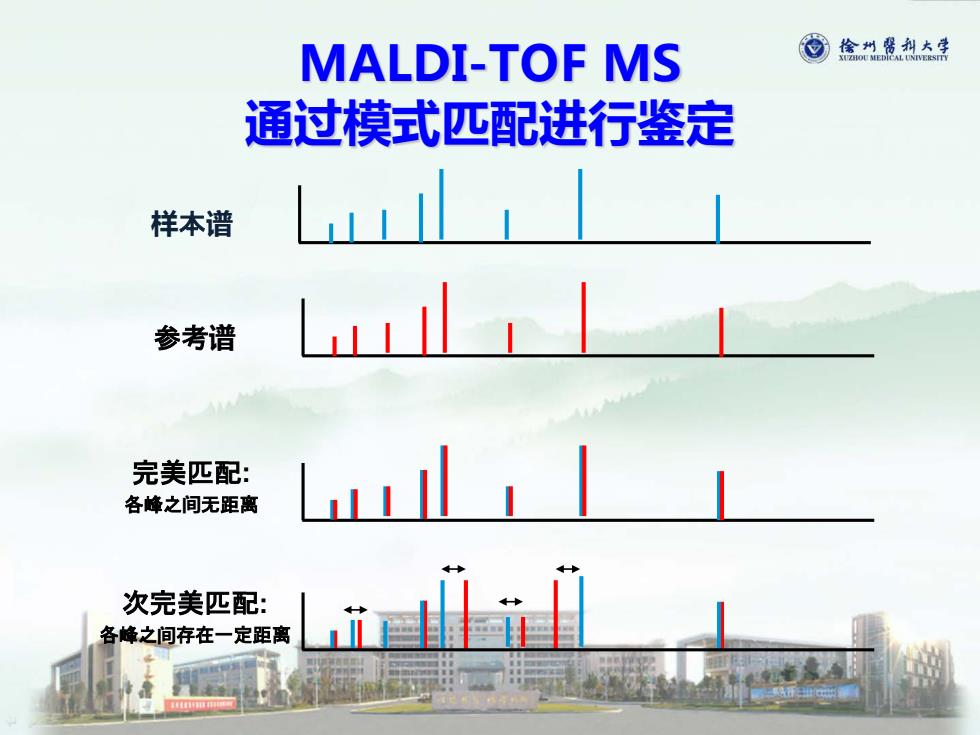
MALDI-TOF MS 偷州号科大手 通过模式匹配进行鉴定 样本谱 参考谱 完美匹配: 各峰之间无距离 次完美匹配: 各峰之间存在一定距离
MALDI-TOF MS 通过模式匹配进行鉴定 样本谱 参考谱 完美匹配: 各峰之间无距离 次完美匹配: 各峰之间存在一定距离
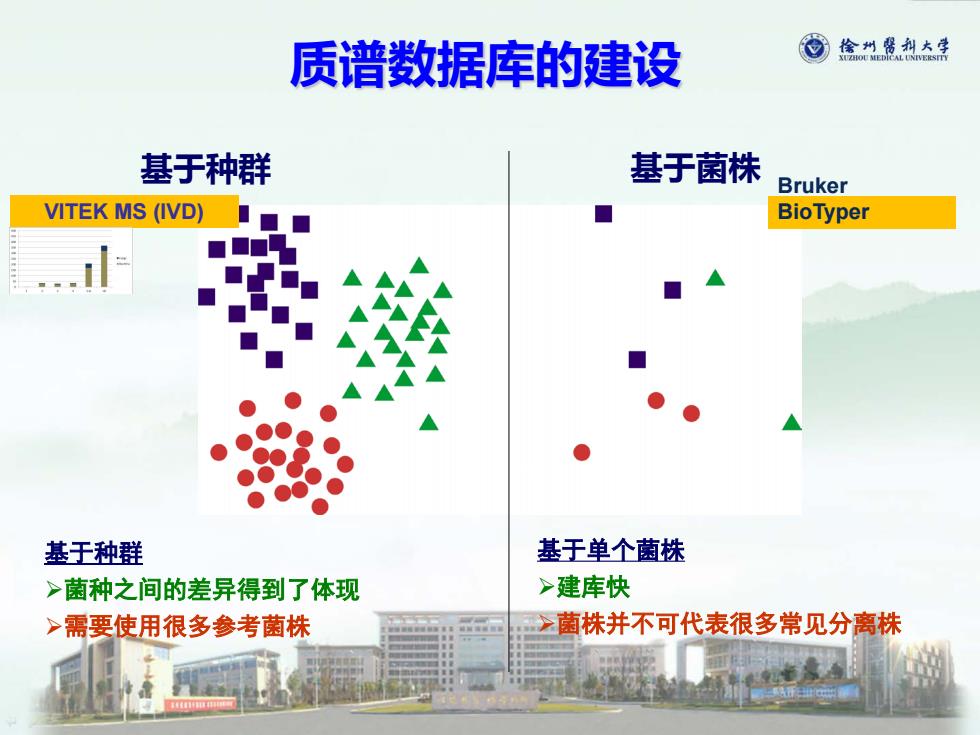
质谱数据库的建设 回徐州号科太手 基于种群 基于菌株 Bruker VITEK MS (IVD) BioTyper 基于种群 基于单个菌株 >菌种之间的差异得到了体现 >建库快 >需要使用很多参考菌株 >菌株并不可代表很多常见分离株
VITEK MS (IVD) 基于种群 基于菌株 Bruker BioTyper 质谱数据库的建设 基于种群 菌种之间的差异得到了体现 需要使用很多参考菌株 基于单个菌株 建库快 菌株并不可代表很多常见分离株
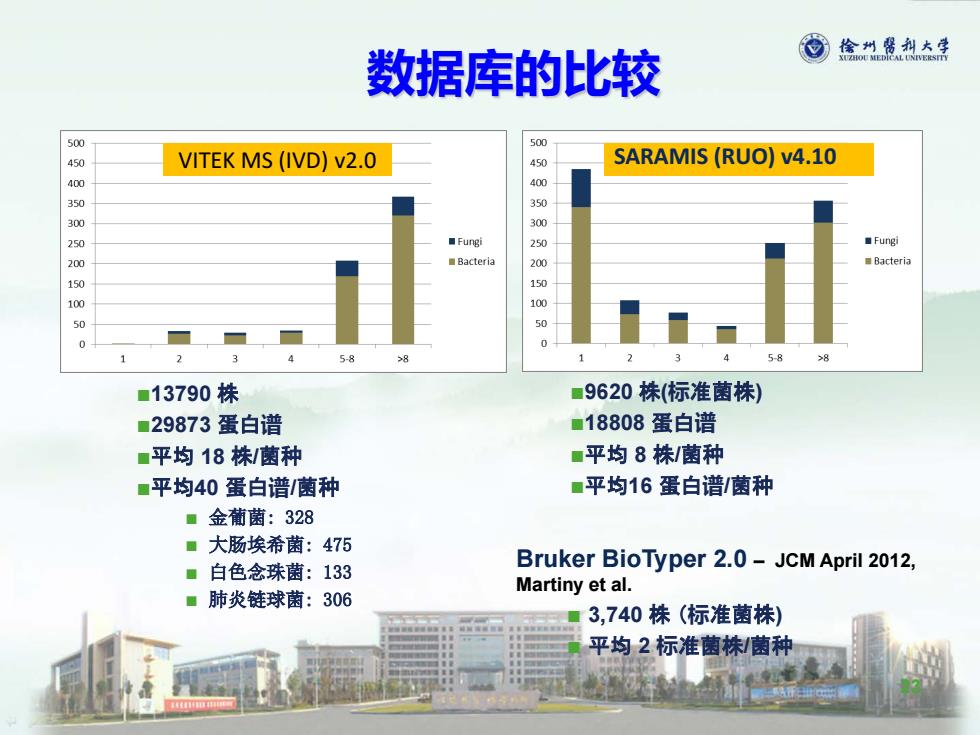
数据库的比较 偷州嘴利太手 500 500 0 VITEK MS(IVD)v2.0 SARAMIS(RUO)v4.10 300 250 ■Fungi 250 ■Fungi 200 ■Bacteria 200 Bacteria 150 150 100 100 0 5-8 1 ■13790株 ■9620株(标准菌株) ■29873蛋白谱 ■18808蛋白谱 ■平均18株/菌种 ■平均8株/菌种 ■平均40蛋白谱/菌种 ■平均16蛋白谱/菌种 ■金葡菌:328 ■大肠埃希菌:475 ■白色念珠菌:133 Bruker BioTyper 2.0-JCM April 2012, ■肺炎链球菌:306 Martiny et al. 3,740株(标准菌株) 平均2标准菌株/菌种
数据库的比较 VITEK MS (IVD) v2.0 SARAMIS (RUO) v4.10 9620 株(标准菌株) 18808 蛋白谱 平均 8 株/菌种 平均16 蛋白谱/菌种 13790 株 29873 蛋白谱 平均 18 株/菌种 平均40 蛋白谱/菌种 金葡菌: 328 大肠埃希菌: 475 白色念珠菌: 133 肺炎链球菌: 306 Bruker BioTyper 2.0 – JCM April 2012, Martiny et al. 3,740 株 (标准菌株) 平均 2 标准菌株/菌种 23

样本准备 ⑤徐州号利太居 1.扫描标本序列号 2. 扫描测试靶板显示空靶点 3.将病原菌放在测试靶板上,并添加基质 扫描VITEK2药敏卡 VITEK MS:pending FDA clearar
样本准备 1. 扫描标本序列号 2. 扫描测试靶板 – 显示空靶点 3. 将病原菌放在测试靶板上,并添加基质 4. 扫描VITEK 2 药敏卡 VITEK MS: pending FDA clearan

检测流程 偷州号利大手 VITEK MS 1.挑取克隆 4.装入质谱仪 2.涂在测试靶板上 3.加基质:1基质(CHCA) a-cyano-4-hydroxycinnamic acid (甲酸酵母菌)
检测流程 1. 挑取克隆 2. 涂在测试靶板上 3. 加基质: 1 µl 基质(CHCA) α-cyano-4-hydroxycinnamic acid (甲酸– 酵母菌) 4. 装入质谱仪