
SAGE 技术的主要理论依据: ❖ 来自转录物内特定位置的一小段寡核苷酸序列 (9~11bp)含有鉴定一个转录物特异性的足够信 息,可作为区别转录物的标签(tag); ❖ 通过简单的方法将这些标签串联在一起,形成大 量多联体(concatemer),对每个克隆到载体的 多联体进行测序,并应用SAGE软件分析,可确定 表达的基因种类,并可根据标签出现的频率确定 基因的表达风度(abundance)
SAGE 技术的主要理论依据: ❖ 来自转录物内特定位置的一小段寡核苷酸序列 (9~11bp)含有鉴定一个转录物特异性的足够信 息,可作为区别转录物的标签(tag); ❖ 通过简单的方法将这些标签串联在一起,形成大 量多联体(concatemer),对每个克隆到载体的 多联体进行测序,并应用SAGE软件分析,可确定 表达的基因种类,并可根据标签出现的频率确定 基因的表达风度(abundance)
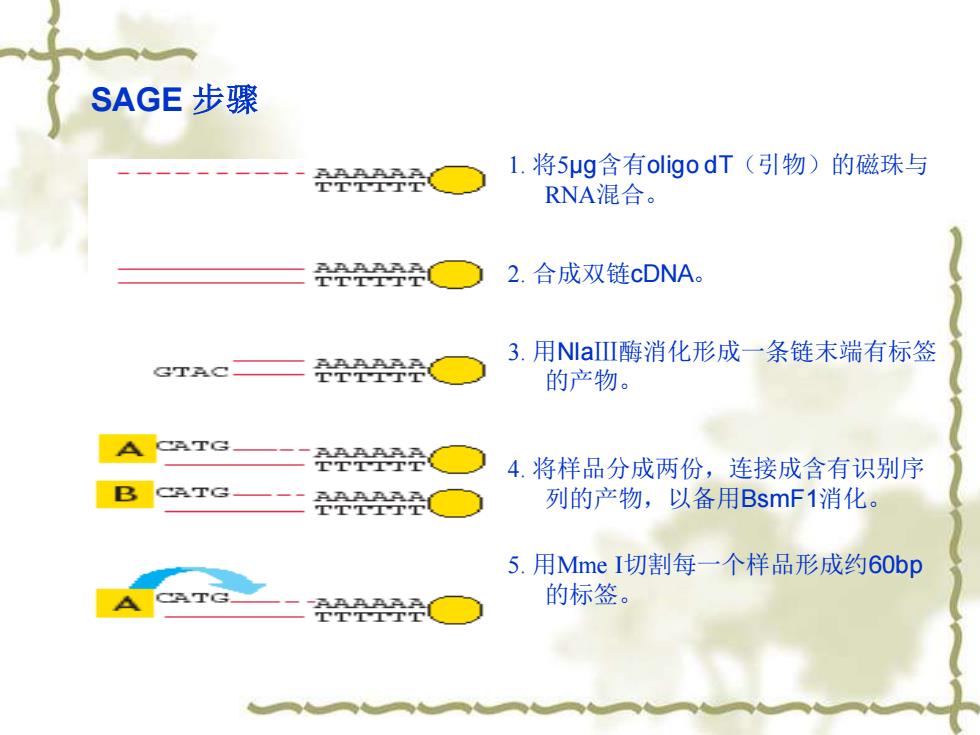
1. 将5μg含有oligo dT(引物)的磁珠与 RNA混合。 2. 合成双链cDNA。 3. 用NlaⅢ酶消化形成一条链末端有标签 的产物。 4. 将样品分成两份,连接成含有识别序 列的产物,以备用BsmF1消化。 5. 用Mme I切割每一个样品形成约60bp 的标签。 SAGE 步骤
1. 将5μg含有oligo dT(引物)的磁珠与 RNA混合。 2. 合成双链cDNA。 3. 用NlaⅢ酶消化形成一条链末端有标签 的产物。 4. 将样品分成两份,连接成含有识别序 列的产物,以备用BsmF1消化。 5. 用Mme I切割每一个样品形成约60bp 的标签。 SAGE 步骤
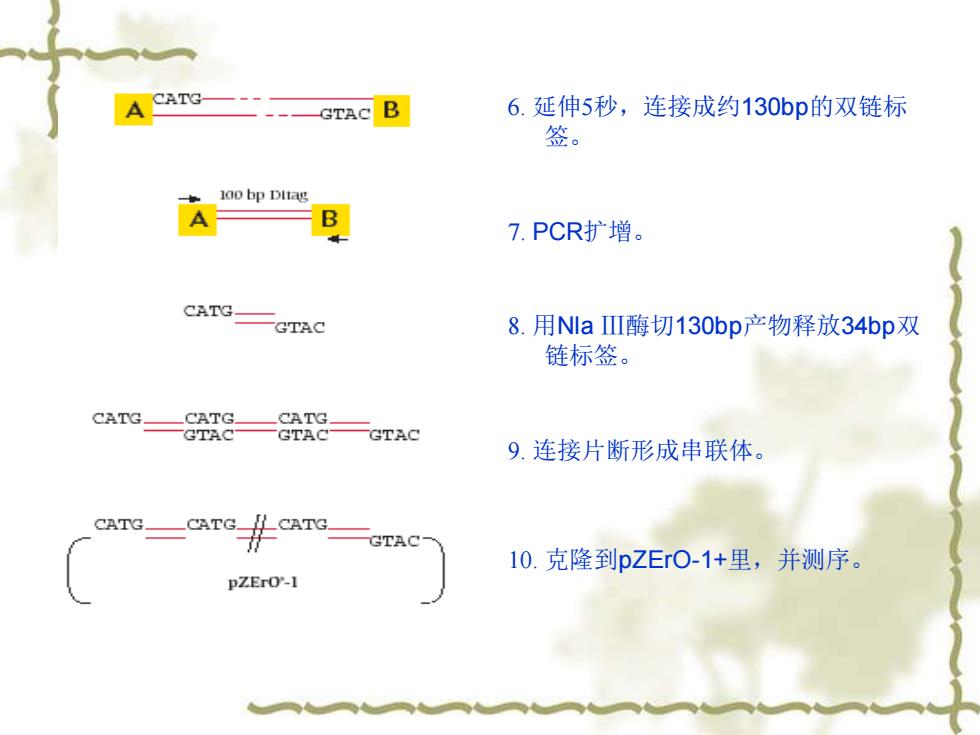
6. 延伸5秒,连接成约130bp的双链标 签。 7. PCR扩增。 8. 用Nla Ⅲ酶切130bp产物释放34bp双 链标签。 9. 连接片断形成串联体。 10. 克隆到pZErO-1+里,并测序
6. 延伸5秒,连接成约130bp的双链标 签。 7. PCR扩增。 8. 用Nla Ⅲ酶切130bp产物释放34bp双 链标签。 9. 连接片断形成串联体。 10. 克隆到pZErO-1+里,并测序
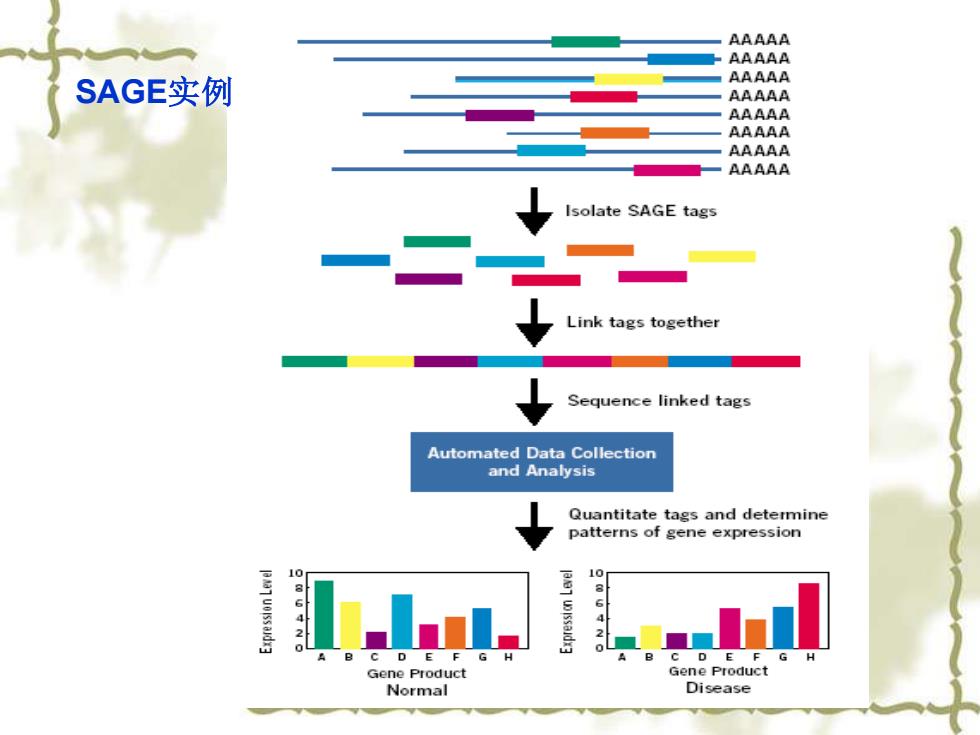
SAGE实例
SAGE实例

5.3 基因芯片 ❖ 何为生物芯片? 生物芯片主要指通过平面微细加工技术在固体芯 片表面构建的微流体分析单元和系统,以实现对 细胞、蛋白质、核酸以及其他生物组分的准确、 快速、大信息量的检测。 它是继大规模集成电路之后的又一次具有深远意义 的科学技术革命
5.3 基因芯片 ❖ 何为生物芯片? 生物芯片主要指通过平面微细加工技术在固体芯 片表面构建的微流体分析单元和系统,以实现对 细胞、蛋白质、核酸以及其他生物组分的准确、 快速、大信息量的检测。 它是继大规模集成电路之后的又一次具有深远意义 的科学技术革命