
虽然不同的启动子序列有所不同,但比较 已经研究过的上百种原核生物的启动子的序列, 发现有一些共同的规律,它们一般长40-60bp, 含A-Tbp较多,某些段落很相似的,有保守性, 称为共有性序列(consensus sequences)。 启动子一般可分为识别(R,recognition).、 结合(B,binding)和起始(l,initiation)三个 区段。 16 2024/12/10
2024/12/10 16 虽然不同的启动子序列有所不同,但比较 已经研究过的上百种原核生物的启动子的序列, 发现有一些共同的规律,它们一般长40-60bp, 含A-T bp较多,某些段落很相似的,有保守性, 称为共有性序列(consensus sequences)。 启动子一般可分为识别(R,recognition)、 结合(B,binding)和起始(I,initiation)三个 区段

转录起始第一个碱基(通常标记位置为 +1)最常见的是A;在-1Obp附近有TATAAT 一组共有序列,因为这段共有序列是 Pr ibnow首先发现的,称为Pr i bnow盒 (Pr ibnow box).;在-35bp处又有TTGACA 一组共有序列。 17 2024/12/10
2024/12/10 17 转录起始第一个碱基(通常标记位置为 +1)最常见的是A;在-10bp附近有TATAAT 一组共有序列,因为这段共有序列是 Pribnow首先发现的,称为Pribnow盒 (Pribnow box);在-35bp处又有TTGACA 一组共有序列
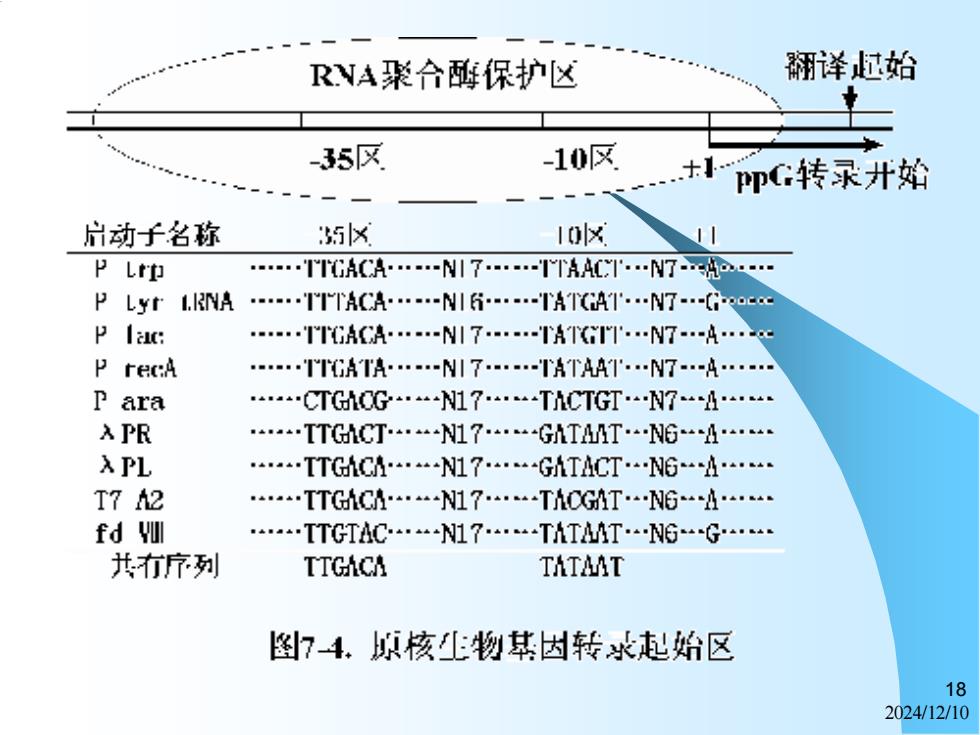
RNA聚合酶保护区 翻译过始 -35区 -10区 +pC转录井始 -一一 启动子名称 35区 1ol区 PL中 .TTGh(CA.N|7 TTAACT.N7o四 PLyr1.lNA.TTTACA.N|行-TATGAT.N7-(Gocc p lac .TTGACA.N|7.TATGIT.N7.A心 p recA .TTGATA.N7-TATAAT.N7.A. P ara .CTGCGN17TACTGT.N7A APR .TTGACT-.-N17 GATANT.N6-A. 入PL .TTGAC-N17.-GATACT.-.N64. T72 .TTGCAN17-ThCG的TN6h fd yl .TTGTAC.N17 TATAAT.-NG-G 共布序列 TTGACA TATAAT 图74.炉核物某因转水起始区 18 2024/12/10
2024/12/10 18

不同的启动子序列不同,与RNA聚合酶的亲 和力不同、启动转录的频率高低不同,即不同 的启动子起动基因转录的强弱不同,例如:P PnP属强启动子,而Pc则是较弱的启动子。 19 2024/12/10
2024/12/10 19 不同的启动子序列不同,与RNA聚合酶的亲 和力不同、启动转录的频率高低不同,即不同 的启动子起动基因转录的强弱不同,例如:PL、 PR、PT7属强启动子,而Plac则是较弱的启动子

(3)操纵区 操纵区(operator)是指能被调控蛋白特 异性结合的一段DNA序列,常与启动子邻近或 与启动子序列重叠,当调控蛋白结合在操纵 子序列上,会影响其下游基因转录的强弱。 20 2024/12/10
2024/12/10 20 (3) 操纵区 操纵区(operator)是指能被调控蛋白特 异性结合的一段DNA序列,常与启动子邻近或 与启动子序列重叠,当调控蛋白结合在操纵 子序列上,会影响其下游基因转录的强弱